|
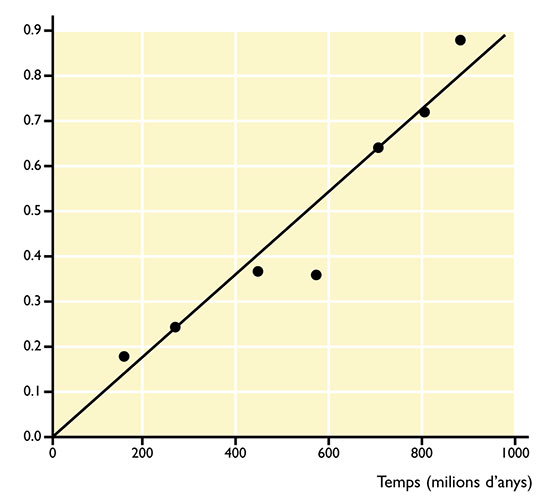
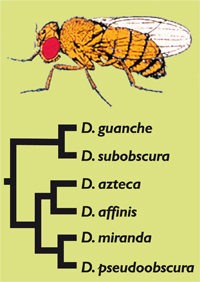
Molecular Evolution: the life’s clock. Molecular evolution studies how molecules change over evolutionary time. This evolution is observable as nucleotide changes in the DNA and as aminoacid changes in the encoded proteins. Molecular evolutionists aim to ascertain the mechanisms involved in the evolution of molecules, and whether the observed changes have been modelled by natural selection or by genetic drift. En sentido amplio se entiende por evolución los cambios que se dan en las características de los organismos en periodos largos de tiempo. Esta definición incluye la dimensión temporal y la noción de cambio. Este cambio tiene que afectar a la información genética, porque son únicamente las características con base genética las que se transmiten de una generación a la siguiente. Cuando en lugar de hablar de evolución en general se habla de evolución molecular, se hace referencia a la evolución de las moléculas, y más concretamente a la evolución de los ácidos nucleicos (que constituyen el material hereditario, y por tanto los genes) y de las proteínas (que son el producto primario de la expresión de estos genes). Comparando la secuencia de aminoácidos de una determinada proteína en especies diferentes, o entre individuos de una misma especie, se detectan cambios (fig.1). Lo mismo sucede si se comparan fragmentos concretos de DNA. La constatación de que a nivel molecular hay diferencias interespecíficas (divergencia) e intraespecíficas (polimorfismo) hace que nos preguntemos por el motivo de los cambios observados y que intentemos distinguir qué factores evolutivos son responsables. Entre estos factores se encuentran la selección natural, que da lugar a las adaptaciones, y la deriva genética, que provoca cambios aleatorios no adaptativos. A menudo se pretende, pues, discernir entre los dos factores, y saber si un cambio determinado es o no adaptativo. Esta pregunta no es nueva, sino que es la misma que se han hecho y se harán los evolucionistas al estudiar niveles de variabilidad más complejos que el de las moléculas. Destino evolutivo de las mutaciones Una nueva variante surgida por mutación puede afectar a la capacidad relativa de supervivencia y reproducción del individuo que la presenta (eficacia biológica). El efecto de este cambio depende del ambiente en el que se encuentra la mutación, tanto biótico como abiótico. Una mutación se considera selectivamente ventajosa si incrementa la eficacia del individuo portador, y selectivamente deletérea si la disminuye. En caso de no afectar a la eficacia biológica, la mutación es selectivamente neutra y su destino no está regido por la selección natural. Imaginemos, por ejemplo, una proteína enzimática que cataliza la degradación de un sustrato determinado con la obtención de energía. Una mutación en el gen que la codifica puede dar lugar a un cambio aminoacídico que le haga perder la capacidad catalítica. Si este sustrato es el único utilizado como fuente de energía, este cambio provocaría la disminución drástica de la capacidad de supervivencia del individuo portador, es decir, la mutación sería selectivamente deletérea. Pero puede ocurrir que una mutación provoque un cambio aminoacídico en la proteína y le posibilite utilizar un nuevo sustrato además del sustrato anterior. Cuando el primer sustrato es escaso y el nuevo sustrato es abundante, el cambio incrementaría la capacidad de supervivencia del individuo portador de la mutación respecto al resto de individuos de la población; sería una mutación selectivamente ventajosa. Finalmente, puede ocurrir una mutación que no provoque ningún cambio aminoacídico en la proteína (o incluso que provoque un cambio aminoacídico que no modifique la capacidad catalítica) y por lo tanto, que no afecte a la capacidad de supervivencia del individuo portador; sería una mutación selectivamente neutra. Las poblaciones de cualquier especie están constituidas por una cantidad finita de individuos. Esto provoca que cualquier cambio surgido por mutación tenga una cierta probabilidad de no estar representado en los individuos de la generación siguiente. Este efecto del azar (deriva genética) es más importante cuanto más reducida sea la cantidad de individuos de la población. El destino de las mutaciones ventajosas (y de las deletéreas) depende fundamentalmente de la selección natural, mientras que el de las mutaciones neutras está regido únicamente por el azar. En principio el destino de una mutación deletérea es la pérdida y, aunque muchas mutaciones ventajosas se fijan por acción de la selección natural positiva, algunas se pueden perder por efecto del azar. En el caso de las mutaciones neutras se ha deducido que la mayoría se pierden en sucesivas generaciones aunque una pequeña fracción se fija. Por lo tanto, entre las mutaciones que se fijan (sustituciones) únicamente están representadas las selectivamente ventajosas y las selectivamente neutras. Figura 1. A) Comparación de secuencias aminoacídicas de un fragmento de la proteína Acp26Aa entre individuos de una misma especie de Drosophila (D. melanogaster) y de una especie diferente (D. Simulans). B) Comparación de secuencias nucleotídicas de una región no codificadora entre individuos de una misma especie de Drosophila (D. subobscura) y de una especie diferente (D. guanche). Un punto indica el mismo aminoácido o nucleótido que la primera secuencia. Reloj molecular y teoría neutralista de la evolución molecular Al comparar la secuencia de una misma proteína de diferentes especies, Zuckerkandl y Pauling constataron que había una relación lineal entre la cantidad de sustituciones aminoacídicas (cambios fijados) entre las parejas de especies y su tiempo de divergencia (fig. 2). Esto significa que por cada proteína la tasa de sustitución (cantidad de sustituciones por residuo aminoacídico por unidad de tiempo) es constante, es decir, cada proteína se comporta como un reloj molecular en el que las sustituciones se producen a un ritmo constante. Este ritmo no puede ser el mismo para diferentes proteínas, aunque sea constante para cada proteína. Esta observación, junto con la detección en los años 60 de niveles elevados de variabilidad proteica intraespecífica, condujo a Kimura a proponer la Teoría Neutralista de la Evolución Molecular (1968). Kimura dedujo que la tasa de sustitución de las mutaciones selectivamente neutras es igual a su tasa de mutación, mientras que la tasa de sustitución de las mutaciones selectivamente ventajosas depende además de otros parámetros como su ventaja selectiva. Sólo en el primer caso (dada la igualdad entre tasa de sustitución y tasa de mutación) es fácil explicar la constancia observada en la tasa de sustitución aminoacídica. Kimura propuso, pues, que la mayoría de los cambios observados en las secuencias de aminoácidos de las proteínas corresponden a mutaciones neutras. La propuesta de Kimura (que posteriormente se hizo extensiva a los cambios observados en el DNA) no significa que la mayoría de mutaciones que se producen sean neutras. Muchas de las mutaciones producidas son deletéreas, y estas mutaciones son eliminadas rápidamente de la población por la selección purificadora. Para poder explicar la constancia observada de las tasas de sustitución, la teoría neutralista propone que una proporción insignificante de los cambios a nivel molecular corresponde a mutaciones ventajosas. Según el neutralismo, la selección purificadora permite explicar los diferentes ritmos del reloj molecular (diferentes tasas de sustitución en diferentes proteínas), ya que la proporción de mutaciones deletéreas puede variar. Diferentes proteínas pueden presentar diferente limitación a variar (limitación funcional), es decir, pueden presentar diferente tolerancia a los cambios aminoacídicos. En aquellas proteínas con gran limitación funcional, la mayoría de cambios alteran la función proteica y disminuyen la eficacia biológica de los individuos portadores (son cambios deletéreos). En contraposición, en aquellas proteínas con menor limitación funcional, la proporción de cambios aminoacídicos que alteran la función (y por lo tanto la de mutaciones deletéreas) es menor. Como, según el neutralismo, la gran mayoría de mutaciones son deletéreas o neutras, hay una relación inversa entre limitación funcional y proporción de mutaciones neutras respecto al total de mutaciones. Las proteínas con mayor limitación funcional tienen una menor tasa de mutación neutra y, por lo tanto, una menor tasa de sustitución. Figura 2. Relación entre el número estimado de sustituciones aminoacídicas en la cadena α de la globina entre parejas de especies de vertebrados y su tiempo de divergencia (adaptado de Kimura, 1983). Las moléculas se utilizan a menudo para reconstruir la historia evolutiva de los organismos (filogenias moleculares) (fig. 3). Si una determinada molécula (proteica o de DNA) presenta una tasa constante de evolución, esta molécula puede ser utilizada como reloj molecular ya que permite estimar el tiempo de divergencia entre especies. Pero las moléculas no siempre evolucionan con una tasa constante, lo que puede afectar tanto a la estimación de tiempo como a la reconstrucción filogenética. La tasa de sustitución de una proteína puede haberse acelerado en un linaje determinado por la fijación de mutaciones selectivamente ventajosas, es decir, por selección positiva. Esta proteína no es un buen reloj molecular y no resultaría conveniente para reconstruir filogenias. Adaptación a nivel molecular y selección natural La selección purificadora elimina aquellas mutaciones que estropean las adaptaciones existentes, ya sean a nivel molecular o a otros niveles. La selección natural positiva es, pues, la única fuerza evolutiva que puede explicar las nuevas adaptaciones. Se ha observado, por ejemplo, que la capacidad del pato asiático Anser indicus de volar por encima de los 9.000 metros está asociada a la elevada afinidad por el O2 de su hemoglobina. La sustitución, promovida por la selección positiva, de una prolina por una alanina en el residuo 119 de la cadena a se encuentra en la base de esta adaptación. Así mismo, se ha establecido que la selección positiva ha tenido un papel importante en la evolución de la insulina en los rumiantes, en la evolución de proteínas implicadas en el reconocimiento óvulo-espermatozoide en invertebrados marinos con fecundación externa, etc. No resulta fácil probar la acción de la selección positiva, y menos aún identificar los cambios moleculares responsables de las nuevas adaptaciones. Dado que la capacidad de adaptación a nuevos retos evolutivos se podría ver limitada por la disponibilidad de mutaciones selectivamente ventajosas, es importante poder establecer si constituyen o no (según los seleccionistas y los neutralistas, respectivamente) una proporción relativamente importante de los cambios moleculares que surgen en cualquier momento evolutivo. Montserrat Aguadé. Dep. de Genètica. Universitat de Barcelona. |
Figura 3. Árbol filogenético de seis especies del género Drosophila.
|