Vigilantes medioambientales
La fauna salvaje, centinela de la contaminación medioambiental por resistencia a los antimicrobianos en Cataluña

El creciente incremento de resistencias a los antimicrobianos (RAM) tanto en seres humanos como en el ganado se atribuye en gran medida al uso excesivo o incorrecto de estos tratamientos. La alarmante aparición de estas resistencias en la medicina humana y veterinaria ha impulsado el desarrollo de diversos estudios de monitorización de los niveles de contaminación RAM en el medio ambiente. En este documento se describe la aparición de genes de resistencia a los antibióticos de último recurso en medicina humana, en una amplia diversidad de animales salvajes. Se sugiere que la fauna salvaje puede ser un buen centinela de la contaminación ambiental por RAM, especialmente en áreas muy pobladas. Por otro lado, los animales salvajes también pueden contribuir a la propagación de bacterias y genes RAM en el medio ambiente y representar un riesgo zoonótico para las personas que estén en estrecho contacto.
Palabras clave: resistencia a los antimicrobianos, genes de resistencia a betalactámicos, Cataluña, genes resistentes a la colistina, fauna salvaje.
En las últimas décadas, la prevalencia de bacterias oportunistas y resistentes a los antimicrobianos (BRAM) asociadas a las infecciones nosocomiales ha aumentado considerablemente en entornos intrahospitalarios. El uso excesivo y el mal uso de los antibióticos en la medicina humana y veterinaria ha provocado la propagación de patógenos resistentes a los antimicrobianos (RAM), lo cual supone un problema de salud global (Pitout y Laupland, 2008). La alarmante aparición de resistencias antimicrobianas en la medicina humana y veterinaria ha impulsado a la realización de diversos estudios para monitorizar los niveles de RAM en el medio ambiente. La implementación urgente de medidas de control basadas en el marco de una sola salud es necesaria para reducir el impacto antropogénico sobre el medio ambiente. Se ha sugerido que la fauna salvaje puede ser un buen centinela de la salud ambiental por su proximidad con las poblaciones humanas, los animales domésticos y los ecosistemas naturales. Pero además, los animales salvajes pueden actuar como portadores asintomáticos de patógenos zoonóticos a largo plazo (Tsiodras et al., 2008), tanto de virus (como la gripe o el virus del Nilo Occidental) como de bacterias (por ejemplo, Salmonella spp., Campylobacter spp. o Staphylococcus aureus resistente a la meticilina) o parásitos (como Cryptosporidium spp.). Por lo tanto, pueden contribuir a propagar estos patógenos entre el ganado y hasta las áreas urbanas, incrementando el riesgo de infección para los animales y los seres humanos.
Las β-lactamasas de espectro ampliado (BLEA) y las β-lactamasas de tipo AmpC (ampC) son las enzimas más comunes que incrementan la resistencia antibiótica a las cefalosporinas de espectro amplio entre las enterobacterias. Estas β-lactamasas (bla) se han diversificado ampliamente en respuesta al uso clínico de medicamentos de nueva generación: cefalosporinas, carbapenémicos y monobactámicos. Las enzimas β-lactamasas se pueden clasificar en tres grupos. El primero de ellos contiene las cefalosporinasas codificadas en el cromosoma de muchas enterobacterias, como ampC, blaCMY, blaACT, blaFOX y blaMIR. Algunas variantes de estas enzimas se han detectado también en plásmidos. El segundo grupo, las serina β-lactamasas, es el de mayor tamaño de espectro amplio contra las penicilinas, cefalosporinas y carbapenémicos. Incluyen las enzimas blaTEM, blaSHV, blaCTX, blaOXA y blaKPC. Estas enzimas están codificadas principalmente en genes de plásmidos que se pueden transferir horizontalmente a diferentes géneros de bacterias. Por último, el tercer grupo, las metalobetalactamasas, dependen del zinc e incluyen las enzimas blaNDM, blaIMP, blaVIM y blaSPM. La aparición de la resistencia a los carbapenémicos –medicamentos de último recurso utilizados en entornos hospitalarios para tratar infecciones graves provocadas por enterobacterias resistentes a los antibióticos betalactámicos– ha comprometido las opciones terapéuticas para la salud humana (Tzouvelekis et al., 2012). Especialmente la variante OXA-48 de las carbapenemasas es cada vez más común entre las infecciones clínicas humanas en Europa (Grundmann et al., 2017).
Otro antibiótico humano de último recurso es la colistina. Los genes móviles de resistencia a la colistina (mcr) son genes de resistencia mediados por plásmidos descubiertos recientemente en humanos y animales destinados a la alimentación (Skov y Monnet, 2016). Se sabe que los genes mcr confieren resistencia al sulfato de colistina –uno de los últimos medicamentos efectivos contra las bacterias gramnegativas multirresistentes (MDR, por las siglas en inglés de multi drug resistance)– (Sun et al., 2018). La colistina se ha utilizado en la medicina veterinaria en las últimas décadas para tratar las infecciones gastrointestinales del ganado, principalmente los cerdos y las aves de corral (EMA, 2016), pero también de forma masiva para la prevención de la colibacilosis en lechones. En consecuencia, el ganado se considera el principal reservorio de selección y propagación de genes mcr a escala global. Hoy en día existe una tendencia mundial a limitar el uso de la colistina en la producción animal por esta gran preocupación en salud pública relacionada con la emergencia y transmisión de genes resistentes (Sun et al., 2018).
Estudios de la resistencia a los antimicrobianos en la fauna salvaje de Cataluña
La propagación de β-lactamasas de amplio espectro se ha estudiado profundamente en las enterobacterias humanas y del ganado, pero todavía necesitamos más estudios sobre su presencia en el medio ambiente y la fauna salvaje. Cataluña cuenta con aproximadamente 7,8 millones de habitantes, con una densidad de población de 242,3 habitantes por kilómetro cuadrado. La comunidad autónoma tiene una gran densidad de granjas de ganado, principalmente porcino, y lidera la producción española de carne de cerdo con un récord de 32,8 millones de animales en 2020, un incremento del 5 % respecto al año anterior, según datos de Eurostat.
En este contexto, los animales salvajes atendidos en el Centro de Recuperación de Fauna Salvaje de Torreferrussa (localizado en los alrededores de Barcelona) han sido objeto de diferentes estudios epidemiológicos. Se examinó a todos los animales con hisopos rectales o cloacales a su llegada al centro, antes de recibir cualquier tratamiento farmacológico o antimicrobiano. La causa de hospitalización más frecuente estaba relacionada con factores antropogénicos relacionados con la persecución directa (heridas de bala, envenenamientos, cautividad ilegal o trampas) o con amenazas humanas involuntarias (colisiones con vehículos, cercados o electrocución). Los métodos de muestreo y los protocolos de gestión de los animales se adecuaron a lo estipulado por el Servei de Fauna (Generalitat de Cataluña) en sus protocolos y principios éticos y a la legislación española.
Aves silvestres
Los primeros estudios realizados revelaron una prevalencia significativa de bacterias resistentes a los antimicrobianos en rapaces salvajes que nunca habían recibido tratamiento antimicrobiano. En concreto, los análisis del 10 % y el 7,4 % de las rapaces salvajes dieron positivo, respectivamente, en salmonela resistente a múltiples medicamentos y en cepas de Campylobacter resistentes a las quinolonas (Molina-López et al., 2011). Entre la gran diversidad de serotipos de salmonela descubiertos en el estudio, destacaba la Salmonella Typhimurium multirresistente DT104, resistente a la ampicilina, el cloranfenicol, la estreptomicina, la sulfonamida y la tetraciclina (ACSSuT), que se encontró en ratoneros comunes (Buteo buteo) y mochuelos (Athene noctua).
En otro estudio, el 6,3 % de rapaces (Bubo bubo, Tyto alba, Strix aluco, Falco tinnunculus, Gyps fulvus) también dieron positivo en salmonela, con una predominancia de la variante S. Typhimurium monofásica 4,12:i:-, cuyo perfil muestra resistencia a la ampicilina, la estreptomicina, las sulfonamidas y la tetraciclina (ASSuT), extensivo a la colistina y el cloranfenicol en algunos casos aislados (Molina-López et al., 2015). En Europa, las cepas monofásicas se suelen caracterizar por un perfil de resistencia ASSuT, y hay pruebas de que los jabalíes (Sus scrofa) son probablemente uno de los reservorios de cepas de este tipo de fago en España (Mateu et al., 2002) y Cataluña (Mejía et al., 2006).
Por otro lado, la frecuencia de aislados de Campylobacter resistentes a las fluoroquinolonas parece responder consistentemente a la presión local selectiva de antimicrobianos. La presencia de cepas aisladas resistentes a las quinolonas en las otras aves salvajes se puede deber a la ingestión de presas. Además, en especies con hábitos alimenticios carroñeros, como el buitre leonado (G. fulvus), la ingestión directa de residuos de antibióticos y bacterias resistentes a los antibióticos de la carroña del ganado o de cadáveres de animales salvajes contagiados podría ser una fuente de infección.
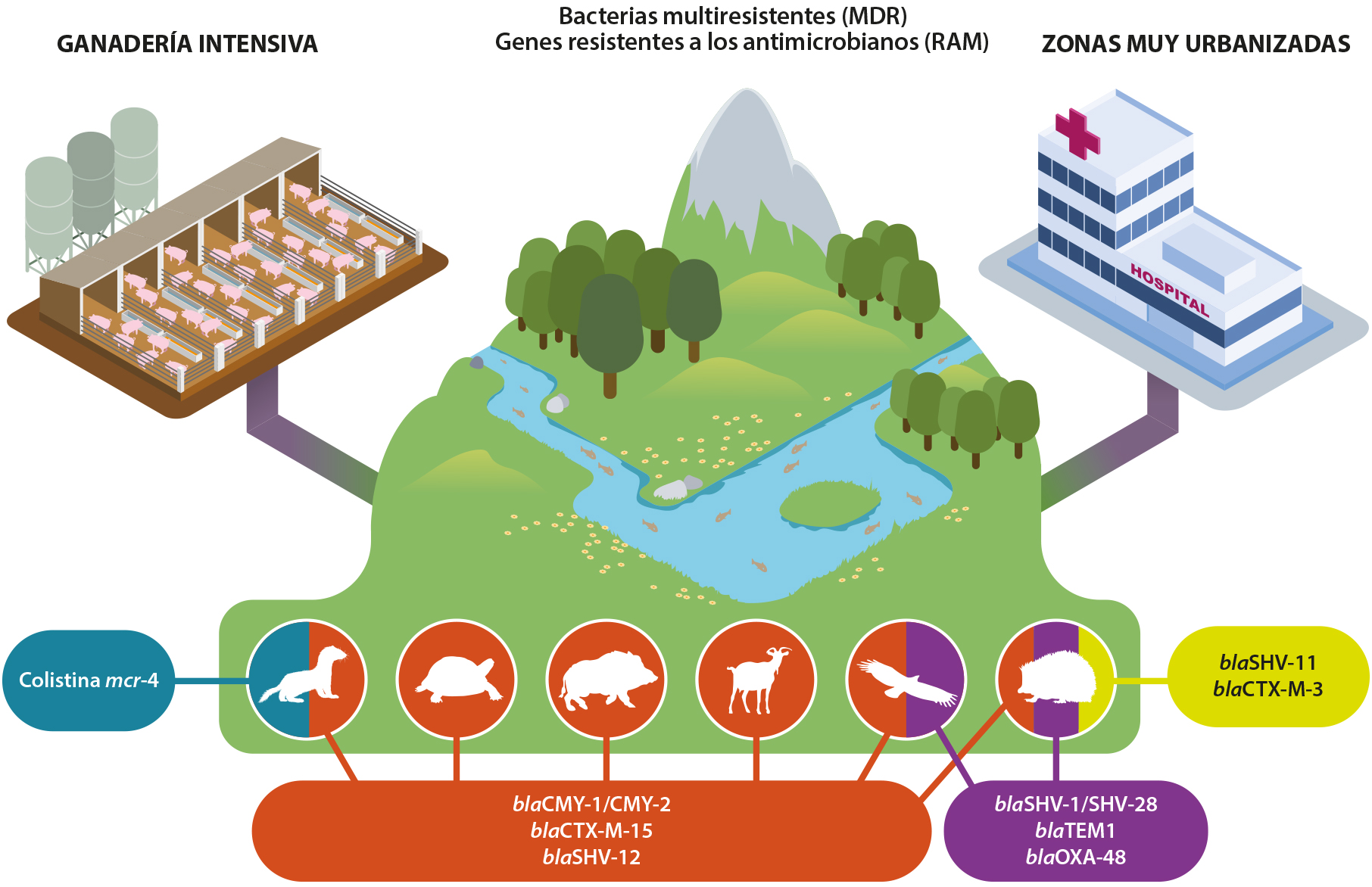
La implementación de análisis de genotipado permitió dar un paso adelante en el estudio de la resistencia antimicrobiana en la fauna silvestre. De esta forma, en 2019 se alertó (por primera vez en España) acerca de la presencia de genes de resistencia a las cefalosporinas en una gran variedad de enterobacterias en mamíferos –principalmente erizos y mustélidos– y aves salvajes –sobre todo rapaces– (Darwich et al., 2019). Significativamente, la prevalencia general de bacterias oportunistas y resistentes a los antimicrobianos en el estudio fue del 13 % (17,3 % en mamíferos salvajes y 11,5 % en aves salvajes). Entre los mamíferos, la mayor prevalencia se encontró en los erizos (13,5 %); entre las aves, fue en las rapaces (7,3 %). La mayoría de BRAM aisladas en esta fauna silvestre contenían una gran diversidad de genes resistentes (50 % blaCMY-2, 23 % blaSHV-12, 20 % blaCMY-1 y 18 % blaCTX-M-15) encontrados habitualmente en las infecciones nosocomiales de los hospitales humanos, como Escherichia coli, Klebsiella pneumoniae, Citrobacter freundii, Enterobacter cloacae, Serratia marcescens y Proteus mirabilis.
Erizos
El erizo común (Erinaceus europaeus) es una especie salvaje común y extendida en Europa. Los erizos son insectívoros de pequeño tamaño, nocturnos y cubiertos de espinas que viven en una amplia variedad de hábitats, generalmente en contacto cercano con los humanos, pero también con el ganado en las zonas rurales. En los últimos años, las poblaciones de erizos ocupan cada vez más zonas con actividad humana, como los jardines de las áreas residenciales o las zonas verdes de las grandes ciudades. Algunas de las consecuencias de esta interacción entre erizos y humanos es que se les suele alimentar, lo que aumenta el riesgo de contacto físico. Por lo tanto, realizamos un estudio con una cohorte de erizos comunes de la misma zona de Cataluña. Los resultados mostraron que un 36,8 % de los especímenes eran portadores de genes RAM, con una especial recurrencia de las bacterias nosocomiales humanas con genes BLEA y AmpC. Además, K. pneumoniae era la bacteria con una mayor proporción de genes de resistencia, seguida de E. coli y C. freundii. Los genes detectados con mayor frecuencia también fueron blaCTX-M-15 (19,3 %), blaSHV-28 (10,5 %), blaCMY-1 (9,7 %), y blaCMY-2 (8,8 %). Es interesante destacar que más de la mitad de las enterobacterias presentaban el fenotipo multiresistente (MDR) con un perfil ampliado de resistencia extendida (XDR) en un 30 % de los aislados. Además, hemos descrito la presencia de genes de la variante de carbapemenasa OXA-48 en enterobacterias de erizos que habitan zonas urbanas muy pobladas de Cataluña (Darwich et al., 2019; Garcias et al., 2021). Como los carbapenémicos no están permitidos en los animales destinados a la alimentación, la creciente presencia de genes que codifican las carbapenemasas en el ganado, las mascotas y los animales salvajes es una alarmante prueba de la contaminación ambiental antropogénica.

Podemos encontrar una posible explicación para la mayor prevalencia de genes de resistencia a los betalactámicos en los erizos en sus hábitos de alimentación y ubicación. Es decir, tiene que estar relacionada con la exposición a aguas residuales o suelos contaminados con bacterias resistentes a los antimicrobianos y residuos de antibióticos. Se ha demostrado que las lombrices, un componente biológico clave del suelo, contribuyen a propagar la resistencia a los antimicrobianos al adquirir antibióticos directamente de la tierra. Por lo tanto, teniendo en cuenta que la mayor parte de la dieta de los erizos consiste en lombrices y otros invertebrados, estos mamíferos pueden verse más expuestos a estas sustancias antibióticas y genes de resistencia que otras especies animales (Garcias et al., 2021).
Jabalíes
Con el mismo objetivo de investigación, se examinó la presencia de E. coli con genes BLEA, así como otros patógenos zoonóticos, en jabalíes (n = 200) que se alimentan en el área metropolitana de Barcelona. Las poblaciones de jabalíes están aumentando a un ritmo constante en todo el mundo. Esto es un motivo de preocupación, porque los jabalíes se han identificado como un reservorio de zoonosis y de patógenos, que reciben a través de su alimentación. Por otro lado, son buenos centinelas para monitorizar el impacto antropogénico del consumo de productos alimentarios en las zonas urbanas. Los resultados de este estudio mostraron que el 8 % de los animales eran portadores de genes de resistencia a las β-lactamasas: blaCMY-2 (3,0 %), blaTEM-1b (2,5 %), blaCTX-M-14 (1,0 %), blaSHV-28 (1,0 %), blaCTX-M-15 (0,5 %) y blaCMY-1 (0,5 %). Además, se detectó Clostridioides difficile toxigénico TcdA+ en dos ejemplares por primera vez en España. Estos resultados indican el gran valor de la especie como centinela de las bacterias resistentes y de agentes zoonóticos en entornos urbanos. De hecho, los jabalíes que se alimentan en localizaciones urbanas y periurbanas estaban más expuestos a fuentes de bacterias resistentes a antimicrobianos que aquellos que habitaban entornos naturales. En concreto, los genes de resistencia betalactámica se aislaron con mayor frecuencia en los jabalíes urbanos y periurbanos (14 % de prevalencia) que en los que habitaban en el bosque (2 % de prevalencia) (Darwich et al., 2021).
Animales semiacuáticos
Los estudios con especies salvajes de aves y mamíferos han mostrado que estos pueden ser reservorios de vectores potenciales para la propagación de bacterias y genes de resistencia a los antimicrobianos (Darwich et al., 2019). Sin embargo, todavía tenemos pocos datos sobre el papel de los animales semiacuáticos como centinelas de la salud ambiental. Por esta razón, el estudio más reciente de nuestro equipo se centró en la presencia de bacterias y genes con resistencia antimicrobiana en animales semiacuáticos de áreas urbanas muy pobladas y zonas de ganadería intensiva de Cataluña, principalmente de producción porcina y avícola (Mengistu et al., 2022).
Se recogieron muestras de especies invasoras como el galápago de Florida (Trachemys scripta) y visón americano (Neovison vison) con hisopos cloacales y rectales. En los resultados, el 55,2 % de los aislados se identificaron como BRAM. E. coli y Pseudomonas fluorescens eran algunas de las bacterias más presentes en los aislados de todas las especies, pero otros agentes nosocomiales como K. pneumoniae, Salmonella spp. o C. freundii también eran comunes. Las pruebas de susceptibilidad fenotípica mostraron que la mayor resistencia era ante los macrólidos y los β-lactámicos. Además, los genes BLEA/Ampc estaban presentes en un 15,4 % de las tortugas y un 8,4 % de los visones americanos. La frecuencia genotípica fue: tetM (20,6 %), blaCMY-2 (13 %), ermB (6,1 %), blaCMY-1 (4,6 %), blaCTX-M-15 (3,1 %) y mcr-4 (0,8 %). Las tortugas tienen una mayor prevalencia de bacterias multiresistentes (MDR) y genes resistentes a antimicrobianos que los mustélidos, aunque el gen de resistencia a la colistina mcr-4 se detectó en los visones. La distribución de genes de resistencia a los β-lactámicos, la eritromicina, la tetraciclina y la colistina se concentraba en las áreas metropolitanas de las grandes ciudades y en regiones con una gran producción porcina. Específicamente, la mayor frecuencia de animales positivos en genes BLEA/ampC se encontró en áreas pobladas de las grandes ciudades (con grandes hospitales) y la mayor frecuencia de animales con genes de resistencia a la tetraciclina y la eritromicina (tetM/emrB) o del gen mcr-4 de resistencia a la colistina se encontró en áreas con una intensa producción ganadera (Mengistu et al., 2022).
En general, se ha informado de la presencia de los genes mcr, principalmente mcr-1 en todo el mundo, en enterobacterias aisladas de humanos, ganado, mascotas, alimentos y fauna salvaje (Skov et al., 2016). Los genes de resistencia a la colistina más frecuentes hallados en enterobacterias porcinas en España son mcr-1 y mcr-4 (Aguirre et al., 2020; Vidal et al., 2020). Por lo tanto, la presencia de mcr-4 en la K. pneumoniae de un visón americano que habite en una de las zonas con la mayor densidad de ganado porcino es un resultado coherente. El abuso del sulfato de colistina en la producción porcina en el pasado como método preventivo para controlar la diarrea tras el destete podría ser la causa de la presión selectiva de bacterias resistentes a la colistina. De hecho, entre 2013 y 2015, España fue el país europeo con mayores ventas de polimixinas para los animales destinados a la alimentación (EMA, 2016), con una prevalencia significativa de los genes de resistencia a la colistina, principalmente mcr-4 (Aguirre et al., 2020). La Agencia Española de Medicamentos y Productos Sanitarios estableció una reducción drástica (97 %) del uso de colistina en cerdos entre 2015 y 2018, con un impacto positivo en la disminución de genes de resistencia a esta (Aguirre et al., 2020). Sin embargo, la presencia de estos genes en aguas naturales sugiere que la reducción en el uso de este antimicrobiano no es suficiente para evitar la selección genética en cerdos vivos o que la contaminación del río proviene de genes mcr estables en purines antiguos no tratados. Por tanto, los animales semiacuáticos son buenos centinelas de la contaminación ambiental (de las aguas, en este caso) por genes y bacterias con resistencia a los antimicrobianos.
Un importante motivo de preocupación
La mayoría de los genes β-lactamasas de espectro amplio detectados en la fauna salvaje catalana también se pueden encontrar con frecuencia en entornos hospitalarios, lo que sugiere que existe una relación entre las bacterias de los animales salvajes y los centros sanitarios (Figura 1). Además, esta prevalencia de bacterias nosocomiales humanas con genes de resistencia a los β-lactámicos en las poblaciones salvajes que viven cerca de estas áreas urbanas es un gran motivo de preocupación, porque estos animales no han recibido nunca tratamiento con antibióticos; es decir, la presencia de genes con resistencia antimicrobiana tiene que estar relacionada con su exposición a aguas residuales o a suelos contaminados o con residuos antimicrobianos. Las plantas de tratamiento de aguas no son del todo eficientes en la eliminación de residuos antibióticos y bacterias resistentes a antimicrobianos (Karkman et al., 2018), y el agua contaminada llega a los ríos y los mares. Además, el estiércol utilizado en los sembrados también los contiene (Han et al., 2021) y enriquece el resistoma tanto del suelo como del agua (Zhou et al., 2020).
La relación entre niveles de resistencia antimicrobiana en el entorno y su presencia en humanos es compleja: existen múltiples combinaciones de antimicrobianos, cepas bacterianas, elementos genéticos móviles (EGM) y especies silvestres, y cada una tiene sus propias dinámicas. Un estudio reciente (Wee et al., 2020) centrado en la relación entre bacterias resistentes en humanos y en el ganado ha mostrado que la transferencia horizontal de genes con resistencia antibiótica mediante EGM como los plásmidos extiende la interconexión entre el humano y el ganado más allá de la transmisión clonal. Es más, la investigación genómica ha revelado genes con resistencia antimicrobiana y elementos móviles muy relacionados en linajes bacterianos humanos y del ganado.
Potencialmente, la secuenciación genómica de alta resolución puede ser una herramienta poderosa que nos ayude a comprender mejor las vías de propagación de bacterias resistentes a antimicrobianos, o cómo se transmite su resistencia de humanos a animales salvajes y viceversa. Esto será clave para desarrollar políticas efectivas de administración antimicrobiana y control de la infecciones para mejorar la salud humana-animal-ambiental.
La urgencia del enfoque de una sola salud
En conclusión, la fauna silvestre asintomática sin ningún registro de tratamiento antibiótico puede ser portadora de bacterias resistentes a los medicamentos humanos o veterinarios y funcionar como reservorios y centinelas de estas bacterias resistentes. La fauna silvestre también puede representar un riesgo zoonótico para el personal de los centros de recuperación de fauna salvaje y para la población general en contacto cercano y continuo con estos animales. En consecuencia, es crucial sensibilizar a la ciudadanía sobre la fuerte interconexión entre hábitats y su relación por múltiples rutas de intercambio, razón por la que los problemas relacionados con la resistencia antimicrobiana han de afrontarse desde una sola salud. Y además, es urgente adoptar este enfoque en regiones densamente pobladas y con una producción ganadera intensiva.
Referencias
Aguirre, L., Vidal, A., Seminati, C., Tello, M., Redondo, N., Darwich, L., & Martín, M. (2020). Antimicrobial resistance profile and prevalence of extended-spectrum beta-lactamases (ESBL), AmpC beta-lactamases and colistin resistance (mcr) genes in Escherichia coli from swine between 1999 and 2018. Porcine Health Management, 6, 4–9. https://doi.org/10.1186/s40813-020-00146-2
Darwich, L., Seminati, C., López-Olvera, J. R., Vidal, A., Aguirre, L., Cerdá, M., Garcias, B., Valldeperes. M., Castillo-Contreras, R., Migura-Garcia L., Conejero, C., & Mentaberre, G. (2021). Detection of beta-lactam-resistant Escherichia coli and toxigenic Clostridioides difficile strains in wild boars foraging in an anthropization gradient. Animals, 11, 1585. https://doi.org/10.3390/ani11061585
Darwich, L., Vidal, A., Seminati, C., Albamonte, A., Casado, A., Lopez, F., Molina-López, R. A., & Migura-Garcia, L. (2019). High prevalence and diversity of extended-spectrum β- lactamase and emergence of OXA-48 producing Enterobacterales in wildlife in Catalonia. PLOS One, 14(8), e0210686. https://doi.org/10.1371/journal.pone.0210686
EMA. (2016). Updated advice on the use of colistin products in animals within the European Union: Development of resistance and possible impact on human and animal health. http://www.ema.europa.eu/docs/en_GB/document_library/Press_release/2016/07/WC500211081.pdf
Garcias, B., Aguirre, L., Seminati, C., Reyes, N., Allepuz, A., Obón, E., Molina-Lopez, R. A., & Darwich, L. (2021). Extended-spectrum b-lactam resistant Klebsiella pneumoniae and Escherichia coli in wild European hedgehogs (Erinaceus europeus) living in populated areas. Animals, 11, 2837. https://doi.org/10.3390/ani11102837
Grundmann, H., Glasner, C., Albiger, B., Aanensen, D. M., Tomlinson, C. T., Andrasević, A. T., Cantón, R., Carmeli, Y., Friedrich, A. W., Giske, C. G., Glupczynski, Y., Gniadkowski, M., Livermore, D. M., Nordmann, P., Poirel, L., Rossolini, G. M., Seifert, H., Vatopoulos, A., Walsh, T., … EuSCAPE working group. (2017). Occurrence of carbapenemase-producing Klebsiella pneumoniae and Escherichia coli in the European survey of carbapenemase-producing Enterobacteriaceae (EuSCAPE): A prospective, multinational study. The Lancet Infectious Diseases, 17, 153–163. https://doi.org/10.1016/S1473-3099(16)30257-2
Han, B., Yang, F., Tian, X., Mu, M., & Zhang, K. (2021). Tracking antibiotic resistance gene transfer at all seasons from swine waste to receiving environments. Ecotoxicology and Environmental Safety, 219, 112335. https://doi.org/10.1016/j.ecoenv.2021.112335
Karkman, A., Do, T. T., Walsh, F., & Virta, M. P. J. (2018). Antibiotic-resistance genes in waste water. Trends in Microbiology, 26(3), 220–228. https://doi.org/10.1016/j.tim.2017.09.005
Mateu, E. M., Martin, M., Darwich, L., Mejia, W., Frias, N., & Garcia Peña, F. J. (2002). Antimicrobial susceptibility of Salmonella strains isolated from swine in Catalonia, Spain. The Veterinary Record, 150, 147–150. https://doi.org/10.1136/vr.150.5.147
Mejía, W., Casal, J., Zapata, D., Sánchez, G. J., Martín, M., & Mateu, E. (2006). Epidemiology of Salmonella infections in pig units and antimicrobial susceptibility profiles of the strains of Salmonella species isolated. The Veterinary Record, 159, 271–276. https://doi.org/10.1136/vr.159.9.271
Mengistu, T. S., Garcias, B., Castellanos, G., Seminati, C., Molina-Lopez, R. A., & Darwich, L. (2022). Occurrence of multidrug resistant Gram-negative bacteria and resistance genes in semi-aquatic wildlife–Trachemys scripta, Neovison vison and Lutra lutra–as sentinels of environmental health. Science of the Total Environment, 830, 154814. https://doi.org/10.1016/j.scitotenv.2022.154814
Molina-López, R. A., Valverdú, N., Martin, M., Mateu, E., Obon, E., Cerdà-Cuéllar, M., & Darwich, L. (2011). Wild raptors as carriers of antimicrobial-resistant Salmonella and Campylobacter strains. The Veterinary Record, 168(21), 565. https://doi.org/10.1136/vr.c7123
Molina-López, R. A., Vidal, A., Obón, E., Martín, M., & Darwich, L. (2015). Multidrug-resistant Salmonella enterica serovar Typhimurium monophasic variant 4,12:i:- isolated from asymptomatic wildlife in a Catalonian wildlife rehabilitation center, Spain. Journal of Wildlife Diseases, 51(3), 759–763. https://doi.org/10.7589/2015-01-019
Pitout, J. D., & Laupland, K. B. (2008). Extended-spectrum beta-lactamase-producing Enterobacteriaceae: An emerging public-health concern. The Lancet Infectious Diseases, 8(3), 159–166. https://doi.org/10.1016/S1473-3099(08)70041-0
Skov, R. L., & Monnet, D. L. (2016). Plasmid-mediated colistin resistance (mcr-1 gene): Three months later, the story unfolds. Eurosurveillance, 21, 30155. https://doi.org/10.2807/1560-7917.ES.2016.21.9.30155
Sun, J., Zeng, X., Li, X., Liao, X., Liu, Y., & Lin, J. (2018). Plasmid-mediated colistin resistance in animals: Current status and future directions. Animal Health Research Reviews, 18, 136–152. https://doi.org/10.1017/S1466252317000111
Tsiodras, S., Kelesidis, T., Kelesidis, I., Bauchinger, U., & Falagas, M. E. (2008). Human infections associated with wild birds. The Journal of Infection, 56, 83–98. https://doi.org/10.1016/j.jinf.2007.11.001
Tzouvelekis, L. S., Markogiannakis, A., Psichogiou, M., Tassios, P. T., & Daikos, G. L. (2012). Carbapenemases in Klebsiella pneumoniae and other Enterobacteriaceae: An evolving crisis of global dimensions. Clinical Microbiology Reviews, 25, 682–707. https://doi.org/10.1128/CMR.05035-11
Vidal, A., Aguirre, L., Seminati, C., Tello, M., Redondo, N., Martín, M., & Darwich, L. (2020). Antimicrobial resistance profiles and characterization of Escherichia coli strains from cases of neonatal diarrhea in Spanish pig farms. Veterinary Sciences, 7(2), 48. https://doi.org/10.3390/vetsci7020048
Wee, B. A., Muloi, D. M., & van Bunnik, B. (2020). Quantifying the transmission of antimicrobial resistance at the human and livestock interface with genomics. Clinical Microbiology and Infection, 26(12), 1612–1616. https://doi.org/10.1016/j.cmi.2020.09.019
Zhou, S., Zhu, D., Giles, M., Daniell, T., Neilson, R., & Yang, X. R. (2020). Does reduced usage of antibiotics in livestock production mitigate the spread of antibiotic resistance in soil, earthworm guts, and the phyllosphere? Environment International, 136, 105359. https://doi.org/10.1016/j.envint.2019.105359