La mayoría de los laboratorios de genética forense utilizan el ADN mitocondrial. El peor problema que tienen que afrontar es el control de la contaminación y la valoración estadística. |
||
|
DNA revolution. Advances in forensic genetics. Analysis of variations in DNA has implied a huge revolution in forensic Medicine. Since Alec Jeffreys introduced the genetic fingerprint in 1985, there has been continuous development in technologies and the type of markers. Microsatellites in autonomic chromosomes are currently the most frequently used markers, but simple nucleotide polymorphisms (SNPs) have emerged as the markers of the future and are already useful for many specific applications, as occurs with polymorphisms in sex chromosomes or in mitochondrial DNA, which are fundamental tools for the forensic investigator. De los grupos sanguíneos a los polimorfismos de ADN nuclear La genética forense es una especialidad que incluye un conjunto de conocimientos de genética necesarios para resolver ciertos problemas jurídicos. Los tipos de pericia más solicitados en el laboratorio de genética forense por los tribunales son casos de investigación biológica de la paternidad, pericias de criminalística biológica (estudio de vestigios biológicos de interés criminal como manchas de sangre, esperma, pelos, etc.) y, finalmente, problemas de identificación. Una de las actividades más importantes del campo son las bases de datos de ADN con fines de identificación criminal. La primera base de datos, y la más amplia, es la del Reino Unido. Contiene unos tres millones de perfiles de ADN y cada día se introducen más de 1.000 perfiles de sospechosos, de convictos o de muestras encontradas en el lugar de los delitos. Antes de la aplicación del ADN, los marcadores genéticos que se utilizaban para estas finalidades (HLA, proteínas, enzimas, grupos sanguíneos) presentaban grandes limitaciones cuando se trataba de analizar muestras degradadas o en minúscula cantidad, algo frecuente en el trabajo forense. Esto era particularmente cierto para el análisis de esperma, manchas de esperma, pelos o cabellos, donde era excepcional proporcionar algún dato acerca de la correspondencia de un vestigio a un presunto agresor, con lo que la ayuda a la justicia era muy limitada. Los polimorfismos de ADN de mayor uso en medicina forense se encuentran en el ADN repetido en tándem y clásicamente se utilizan los denominados minisatélites y microsatélites que consisten en repeticiones de fragmentos de ADN de número variable. Las repeticiones en el ADN microsatélite son de tamaño pequeño (de 2 a 6 pares de bases o bp) por lo que se suelen denominar STR (Short Tandem Repeats). Poniendo un ejemplo, un STR puede tener una estructura como ACTT ACTT ACTT ACTT ACTT ACTT ACTT ACTT… hasta un número n de repeticiones. Los individuos nos diferenciamos por el número de repeticiones de esa secuencia. Un individuo 8-12 para ese STR significa que tiene 8 veces la unidad de repetición (ACTT) en un lugar específico de un cromosoma (locus génico) y 12 veces en el locus correspondiente del cromosoma homólogo. Los minisatélites y microsatélites, además de ser extraordinariamente polimórficos (esto es, variables entre los individuos de una población), poseen una herencia mendeliana simple. Esto significa que el individuo 8-12, que antes pusimos de ejemplo, ha heredado uno de los alelos de su madre y otro de su padre biológico. En el campo forense utilizamos básicamente STR de 4bp y 5bp en la unidad de repetición. Los de menos repeticiones son muy propensos a artefactos (bandas tartamudas) lo que dificulta la interpretación de perfiles de ADN obtenidos a partir de mezclas de diferentes individuos. En 1984, el genetista británico Alec Jeffreys y su grupo descubrieron la enorme variación que tienen los minisatélites entre los individuos y acuñaron el término «DNA fingerprint» (huella genética de ADN) y realizaron las primeras aplicaciones forenses. El análisis de minisatélites mediante sondas se abandonó recientemente por algunas dificultades en su empleo forense, ya que es muy difícil analizar ADN degradado o en pequeña cantidad con sondas y esto dificulta gran parte de las aplicaciones forenses. Por fortuna esto fue solucionado con la aparición de la reacción en cadena de la polimerasa y el descubrimiento de los microsatélites. El primer sistema analizado por PCR con fines forenses fue un polimorfismo de ADN codificante de la región HLA: el locus HLA DQA1, que se detectaba mediante sondas que reconocen cada alelo del sistema previamente fijadas a una membrana. Posteriormente se utilizaron minisatélites de pequeño tamaño, pero fue el descubrimiento de los microsatélites o STR lo que abrió enormes posibilidades en este campo. Sus ventajas eran notables, ya que ofrecían junto a pequeños tamaños (y por lo tanto más resistencia a la degradación), un buen poder de discriminación y facilidades para ser amplificados de forma simultánea con PCR múltiplex (esto es, amplificar varios sistemas STR simultáneamente a partir de la misma muestra). El análisis de los productos amplificados se ha facilitado en gran medida gracias al uso de fluorocromos y sistemas automatizados (secuenciadores automáticos de ADN) que permiten la visualización de varios mini o microsatélites simultáneamente. Los polimorfismos analizables por PCR antes de ser aceptados para la práctica forense deben cumplir una serie de requisitos y pasar sucesivos controles de validación. Actualmente se suelen analizar hasta 15 STR (estandarizados y validados) a partir de la misma muestra biológica utilizando secuenciadores automáticos y PCR múltiplex. Los múltiplex comerciales de Applied Biosystems (Identifiler) y Promega (PowerPlex 16) que contienen 15 STR y el marcador de sexo amelogenina son los más utilizados. |
«Los tipos de pericia más solicitados por los tribunales son casos de investigación biológica de la paternidad, pericias de criminalística biológica y problemas de identificación» | |
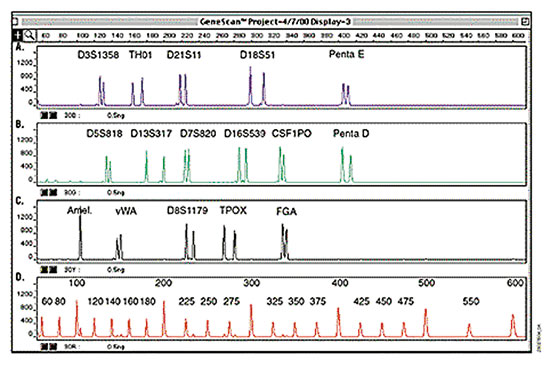
Múltiplex de STR utilizando el equipo comercial PowerPlex16 (Promega) que incluye quince STR y el marcador de sexo amelogenina marcados con diferentes fluorocromos. En la calle final se ve un estándar interno. |
«El descubrimiento de los microsatélites o STR fue lo que abrió unas enormes posibilidades en el campo de la genética forense» | |
|
Para análisis de criminalística biológica se prefiere usar STR con un pequeño tamaño en el producto amplificado (menos de 200 bp) pues el tamaño es inversamente proporcional a la degradación y en muestras muy degradadas sólo cabe esperar éxito con la amplificación de estos sistemas. Actualmente se han diseñado numerosos múltiplex disponibles comercialmente con este tipo de STR que se suelen denominar miniSTR. También son muy interesantes las repeticiones de 5 nucleótidos (pentanucleotide repeats) cuando se trata de analizar muestras mezcladas con diferentes individuos pues tienen menos artefactos que otros STR. En varios múltiplex disponibles comercialmente se incluyen este tipo de STR. Los polimorfismos de ADN mitocondrial Hasta no hace muchos años, el estudio de los polimorfismos de ADN se había centrado mayoritariamente en el análisis de marcadores nucleares. Sin embargo, durante estos últimos años, el interés por este genoma mitocondrial ha crecido considerablemente. El ADN mitocondrial (ADNmt) posee múltiples aplicaciones en el campo de la genética forense debido fundamentalmente al modo de herencia que presenta, la elevada tasa de mutación y a la existencia de miles de moléculas por célula, lo que permite estudiarlo en condiciones en las que el material biológico a analizar se encuentra en mal estado o en cantidad insuficiente para estudiar cualquier otro marcador nuclear. El ADNmt humano es una molécula de ADN circular, cerrado y de doble cadena y relativamente pequeño, ya que mide 16.569 bp y fue secuenciado en su totalidad por primera vez en 1981 por Anderson y colaboradores. Se hereda prácticamente sólo por vía materna y, aunque representa menos del 1% del ADN celular total, posee un gran número de copias. Se estima que las células de mamíferos contienen varios miles de copias de ADNmt dependiendo del tipo de tejido. Esta característica es la que le confiere más posibilidades de éxito en muestras degradadas. Hay dos regiones principales en el genoma mitocondrial: una gran región codificante (90%) y una región pequeña de aproximadamente 1.2 kb conocida como «región control». Esta región es muy polimórfica y contiene dos regiones hipervariables bien caracterizadas, conocidas como la región hipervariable I (HVI) y la región hipervariable II (HVII), de aproximadamente 400 bp cada una. Todos estos elementos de control hacen que esta región no sea en conjunto selectivamente neutra. La identificación forense mediante ADNmt se basa fundamentalmente en el estudio de esta región no codificante. De la misma manera, dada su alta variabilidad, es de gran utilidad en el estudio antropológico de la evolución humana ya que las diferencias interpoblacionales a nivel mitocondrial son un reflejo molecular de acontecimientos históricos que se han venido sucediendo durante los últimos milenios. Toda la molécula de ADNmt ha sido secuenciada en toda su longitud y la numeración de la molécula del ADNmt es aquella establecida por Anderson y colaboradoresen 1981. Empieza arbitrariamente cerca del punto medio de la región control, de forma que esta región se extiende desde la posición 16024 a la 16569 y luego se continúa desde la 1 hasta la 576. Las dos regiones hipervariables se extienden aproximadamente desde las posiciones 16024 a la ~16365 HVI y desde la 73 hasta la ~340 (los límites no están en cualquier caso rígidamente establecidos y varían según los estudios). El ADNmt evoluciona de 5 a 10 veces más rápidamente que un segmento medio del ADN nuclear y una de sus consecuencias es la heteroplasmia, esto es, una mezcla intracelular de moléculas mutantes y normales que puede dificultar la interpretación forense pero también proporcionar un dato de interés en la identificación. También está cobrando un valor cada vez mayor el estudio de variaciones nucleotídicas simples en región codificante (SNP) del ADNmt. Estas variaciones permiten definir con mayor precisión el haplogrupo al que pertenece una muestra, lo que aumenta su valor identificativo. Por ejemplo, un porcentaje importante de las muestras europeas pertenecen al haplogrupo H (esto es, a un mismo linaje) sin posibilidad de diferenciar unos individuos de otros si no se recurre al análisis de SNP de región codificante, que aumenta sensiblemente el poder del test. El ADNmt es importante en genética forense en dos circunstancias: en el análisis de pelos y cabellos y en el análisis de muestras degradadas. Por su mejor comportamiento en muestras degradadas el ADNmt es esencial para muchos casos de identificación a partir de restos óseos. Se han obtenido secuencias de ADNmt en muestras de miles de años y la prueba ha servido para solucionar numerosos enigmas históricos como la identificación de los restos de la familia Romanov, los últimos zares de Rusia, asesinados durante la revolución bolchevique de 1918. Qué había pasado con los restos era un misterio y numerosas leyendas, como la de la princesa Anastasia, circulaban y aún circulan por todo el mundo. Un grupo de espeleólogos, geólogos y médicos hallaron los restos de la familia imperial en 1991. En 1998, y gracias a las pruebas demostradas con el ADN, se supo que todos los cadáveres encontrados pertenecían a la familia imperial. Los siete componentes eran, sin dudarlo, el zar Nicolás II, su esposa y todos sus hijos. Para la confirmación pidieron muestras de ADN a diferentes miembros de la realeza europea más cercana a los Romanov, a miembros de las casas reales danesa, británica y a la griega. El ADN mitocondrial fue decisivo en este caso para la resolución del caso. La mayoría de los laboratorios de genética forense utilizan hoy día el ADNmt; el mayor problema que plantea es el control de la contaminación y la valoración estadística. La Sociedad Internacional de Genética Forense (ISFG) ha publicado recomendaciones estrictas para que se haga un uso apropiado. La valoración estadística exige que se disponga de bases de datos poblacionales muy amplias (es decir, perfiles de ADNmt totalmente anónimos con el fin de estimar la frecuencia) y en este sentido se está haciendo un esfuerzo colaborativo importante a escala mundial. Los polimorfismos del cromosoma Y A pesar de representar sólo el 2% del componente cromosómico humano, el cromosoma Y posee unas características que lo diferencian del resto de los cromosomas y le confieren gran utilidad desde el punto de vista tanto forense como antropológico. Una de las características más importantes es que la mayor parte del cromosoma Y no recombina durante la meiosis. Esto determina que todas las secuencias localizadas en esta región no pseudoautosomal son heredadas como un bloque de padres a hijos, constituyendo un grupo de ligamiento. La única fuente posible de variación se produce por eventos mutacionales. Otra de las características del cromosoma Y es que presenta una baja diversidad, a pesar de esto se han descrito numerosos polimorfismos. De todos ellos los más interesantes a efectos forenses son los microsatélites, aunque los marcadores bialélicos (mayoritariamente SNP) están empezando a tener una gran importancia por su sensibilidad para poder ser analizados en muestras mínimas y por la posibilidad de definir con precisión el haplotipo y poder así dar datos sobre el posible origen geográfico de una muestra. Actualmente se pueden encontrar en bases de datos genómicas centenares de STR de cromosoma Y pero también se ha estandarizado su uso. Así, los STR que integran el llamado «haplotipo mínimo» (DYS19, DYS385, DYS389I, DYS389II, DYS390, DYS391, DYS392 y DYS393) son usados por la mayoría de los laboratorios forenses en la actualidad y existen múltiplex comerciales ampliamente usados. |
«El cromosoma Y posee unas características que lo diferencian del resto de los cromosomas y le confieren gran utilidad desde el punto de vista forense»
«Los avances tecnológicos en el área forense suelen tener una aplicación relativamente lenta por la necesidad de validación previa» |
|

El ADN mitocondrial se ha convertido en la clave para que los laboratorios puedan analizar pelos y muestras degradadas. Por este motivo ha sido esencial para la identificación a partir de restos óseos. En la imagen, proceso de extracción y ampliación de muestras críticas en el Instituto de Medicina Legal de Santiago de Compostela. Pese a la existencia de robots para la extracción de muestras de ADN, éstos no están preparados todavía para obtener las muestras críticas. |
«La prueba de ADN mitocondrial ha servido para solucionar numerosos enigmas históricos como la identificación de los restos de la familia Romanov» | |
|
Los microsatélites localizados en el cromosoma Y han irrumpido con gran fuerza en el panorama de los marcadores genéticos de uso forense, debido a que suponen una ayuda inestimable para ciertas situaciones forenses específicas como son algunos casos de investigación de la paternidad difíciles y especialmente casos criminales con mezcla de ADN masculino y femenino. Así, los polimorfismos de cromosoma Y son importantes en el análisis de muestras en delitos contra la libertad sexual, en las que el esperma u otras células del agresor están mezcladas con células femeninas de la víctima, ya que si son utilizados marcadores autosómicos se obtiene una amplificación preferencial del mayor de los componentes (usualmente ADN femenino), que enmascara el perfil genético del asaltante. Además de los microsatélites, cada vez está siendo más importante el uso de polimorfismos nucleotídicos simples (SNP) de cromosoma Y. Estos completan la información de los STR y son útiles para conocer el origen geográfico de la persona que dejó la muestra. La Sociedad Internacional de Genética Forense (ISFG) ha publicado recientemente recomendaciones para el uso correcto de estos polimorfismos, su nomenclatura y especialmente la valoración estadística de los resultados, que comparte los problemas del ADNmt. Como en éste, existe la necesidad para los polimorfismos de cromosoma Y de grandes bases de datos poblacionales para estimar la frecuencia de los haplotipos y se ha realizado un esfuerzo colaborativo a nivel mundial muy importante en este sentido. STR y SNP de cromosoma X también están siendo cada vez más usados en la resolución de casos de paternidad difíciles como herramientas complementarias a todas las anteriores. Los métodos de futuro A diferencia de otros campos de la genética, los avances tecnológicos en el área forense suelen tener una aplicación a la realidad pericial relativamente lenta por la necesidad de validación previa y de incorporación a programas de control de calidad. Los polimorfismos de más futuro son los más simples, es decir, los SNP de cromosomas autosómicos, que poseen una tasa de mutación muy baja que los hace idóneos para pruebas de paternidad. Frecuentemente, debido a la alta tasa de mutación de los STR, en las pruebas de paternidad aparecen inconsistencias que parecen exclusiones pero que son mutaciones. Al incluirlas en los cálculos estadísticos, la probabilidad de paternidad baja drásticamente. El uso de paneles de SNP está solucionando todos esos casos. Además, está demostrando ser particularmente útil en los casos de paternidad con relaciones familiares en el grupo (por ejemplo, que los posibles padres sean dos hermanos), así como en paternidades que se han de realizar a partir de material degradado (por ejemplo, tras la exhumación de los restos óseos del presunto padre). Dado que el tamaño de los productos amplificados puede ser mínimo, la eficacia de los SNP en material degradado y en bajo número de copias supera con creces a los STR. Su simplicidad los hace susceptibles de análisis a gran escala y particularmente de análisis con chips o microarrays de ADN. Entre todos los tipos de nuevas tecnologías quizá los formatos basados en MALDITOF MS (Matrix Assisted Lasser Deionization Time-Of-Flight Mass Spectrometry) son los de mayor futuro, sin olvidar formatos electroforéticos basados en minisecuenciación. Así, la mayor parte de los laboratorios forenses avanzados que los han incorporado en la rutina emplean SNaPshot (una tecnología basada en minisecuenciación y electroforesis) y GenPlex (una tecnología que combina OLA –una técnica de aplicación basada en ligasas– y PCR). El número de SNP que se necesitan a efectos forenses es relativamente bajo comparado con otras aplicaciones de los SNP en genética humana. Unos 50-60 SNP de frecuencias equilibradas tienen aproximadamente el mismo nivel de discriminación que 12 STR. El grupo SNPforID ha validado un set de 52 SNP autonómicos que está siendo muy empleado, así como diversos paquetes de SNP para otras aplicaciones forenses de los SNP. En este sentido, una de las aplicaciones más interesantes es el uso de marcadores informativos de ancestros (AIM) para predecir el origen geográfico de la persona que ha dejado una muestra biológica. Este tipo de prueba ha sido empleada con éxito en los atentados del 11-M de Madrid para predecir el origen geográfico de perfiles no identificados encontrados en objetos importantes para la investigación judicial del caso. La eficacia del método es muy alta, hasta el punto de poder predecirse con una elevada probabilidad en muchos casos si una muestra es sureuropea o norteafricana, dos poblaciones tan próximas geográficamente y con una larga historia compartida. Los SNP son también importantes y lo serán aún más en el futuro para predecir características físicas de un individuo a partir de una muestra con fines de investigación policial. Así, a partir de un vestigio, ya se puede decir si una persona tiene el pelo pelirrojo y se está haciendo una investigación muy intensa en otras características físicas como el color de los ojos, de la piel, lateralidad, rasgos faciales, etc. ADN en bajo número de copias y mezclas de perfiles de ADN La sensibilidad de las técnicas actuales ha hecho posible que se puedan obtener perfiles de ADN a partir de sólo unas pocas copias (agarrar una prenda o un objeto unos pocos segundos ya es suficiente para dejar un perfil). Por desgracia esto ha hecho más compleja la interpretación al aumentar el número de casos en los que existe una mezcla de perfiles de ADN. El análisis de mezclas de ADN es pues una práctica habitual en la rutina forense y es uno de los mayores retos. La interpretación es delicada, dado que se hace imposible diferenciar qué alelos pertenecen a cada contribuyente a la mezcla. Si además la mezcla está desequilibrada (uno de los contribuyentes aportó más cantidad de fluido biológico que el otro u otros) podemos cometer errores al asignar alelos que en realidad son artefactos de la amplificación (sttuters) o al no detectar alelos que realmente existen en la mezcla (drop out alélico). Para solucionar los problemas de interpretación de mezclas de perfiles se han creado programas informáticos que nos ayudan a interpretar mezclas (Pendulum) cuando no existe la posibilidad de la separación física de los perfiles genéticos. Por otro lado, en casos de delitos contra la libertad sexual, donde la separación física es posible, es cada vez más frecuente el uso de microscopios capturadores de espermatozoides. Estos microscopios permiten aislar mediante láser cada espermatozoide que visualizamos e introducirlo en un tubo para extraer su ADN independientemente del de la víctima, evitándose así los perfiles mezcla. Automatización y rapidez del análisis forense Uno de los principales problemas hoy en los laboratorios forenses es el gran número de datos electrónicos que se generan regularmente. Desde que una muestra llega al laboratorio hasta que se escribe el informe pericial son muchos los pasos que se dan y los resultados analíticos que se producen. Hemos de tener en cuenta el carácter judicial de las muestras que analizamos, y como tales han de estar controladas en todo momento, siguiendo una cadena de custodia tanto de los efectos como de las sub-muestras que se generan de ellos. Por tanto, en los últimos años ha sido necesario desarrollar sistemas informáticos que permitieran el manejo fácil y seguro de toda esta información generada, los LIMS (Laboratory Information Management Systems). Estos sistemas nos permiten saber dónde están en cada momento las muestras que se analizan y en qué fase del análisis se encuentran (trazabilidad), nos proporcionan las herramientas necesarias para las revisiones administrativas de cada caso y contienen módulos analíticos para estudios de ADN (comparación y almacenamiento de perfiles genéticos, análisis estadísticos, etc). Las técnicas de análisis en sí también están automatizándose. Existen hoy en día robots para extraer ADN de un elevado número de muestras en un par de horas, aunque aún no están preparados para muestras críticas. Los sistemas miniaturizados compactos (lab-on-a-chip) están actualmente en fase de diseño y experimentación. Han sido de muy difícil desarrollo al basarse aún actualmente en la identificación en STR, lo que exige recorridos electroforéticos de cierta distancia, pero han cobrado un nuevo auge con la potencial aplicación de los SNP. En el futuro próximo se dispondrá de sistemas que se puedan llevar a la escena del crimen y estén conectados de forma directa con bases de datos. En algunos países, dada la insuficiencia actual de este tipo de aproximaciones, se utilizan laboratorios móviles que se llevan a la escena del crimen para tratar de minimizar el tiempo de respuesta. BIBLIOGRAFÍA Angel Carracedo. Instituto de Medicina Legal, Universitat de Santiago de Compostela. |
«El uso de marcadores informativos de ancestros ha sido utilizado con éxito en el análisis de los atentados del 11-M de Madrid» | |