Esquivando balas mágicas
La evolución de la resistencia a antibióticos en las bacterias
Las bacterias resistentes a antibióticos representan uno de los principales problemas actuales de salud pública y predicciones recientes indican que pronto se convertirán en la primera causa de mortalidad a nivel mundial. El funesto recorrido desde la introducción de los antibióticos en la práctica clínica hasta la actual amenaza de una era postantibiótica se ha transitado en tan solo unas pocas décadas. Así, la evolución de la resistencia a los antibióticos es probablemente el ejemplo más espectacular de evolución de innovaciones en un sistema biológico que hemos podido observar en tiempo real. En este artículo discutimos algunas de las claves evolutivas y moleculares que han permitido a las bacterias recorrer este camino.
Palabras clave: innovación evolutiva, resistencia a antibióticos, bacterias, plásmidos, transferencia genética horizontal.
Introducción
La capacidad de los seres vivos para explotar nuevas oportunidades ecológicas depende de su habilidad para innovar. La evolución de innovaciones consiste en la adquisición de nuevos caracteres que permiten a los organismos expandir su nicho y afrontar nuevos retos ambientales. Algunos ejemplos clásicos de innovación evolutiva son la aparición de las flores en las plantas o de la capacidad para volar en los animales (Mayr, 1963). En muchos casos estas innovaciones son obvias a nivel fenotípico, incluso espectaculares; solo hay que levantar la vista y observar una bandada de aves cruzando el cielo para comprobarlo. Sin embargo, las bases moleculares que subyacen a estas adaptaciones no son tan evidentes. ¿Cómo se generan las innovaciones? ¿Cuáles son las bases genéticas de este proceso? Estas preguntas han sido objeto de estudio durante décadas y continúan abiertas a pesar de todo lo aprendido al respecto (para más información sobre el origen de las innovaciones, véase Wagner, 2015). En este artículo vamos a intentar ilustrar algunas de las claves del proceso de innovación evolutiva a través de uno de los ejemplos más dramáticos de los que disponemos actualmente: la evolución de resistencia a antibióticos en las bacterias.
«700.000 personas mueren cada año en el mundo como consecuencia de infecciones resistentes a los antibióticos»
Una advertencia desatendida
Los antibióticos son sin lugar a duda uno de los descubrimientos de mayor relevancia en la historia de la humanidad. Estas «balas mágicas», como las definió Paul Ehrlich ya a principios del siglo xx (Tan y Grimes, 2010), son moléculas capaces de inhibir específicamente el crecimiento bacteriano y han supuesto la base para el desarrollo de la medicina y la cirugía modernas. Y es que, de hecho, parecen balas mágicas; los antibióticos son increíblemente eficientes en el tratamiento de infecciones bacterianas. Paradójicamente, esta gran efectividad ha sido también el origen de su declive, ya que durante décadas se ha producido un uso desmesurado y en muchos casos inadecuado de este preciado recurso, lo cual ha promovido la evolución de la resistencia a antibióticos en las bacterias. Este fenómeno no debería sorprendernos, puesto que, en su discurso de aceptación del Premio Nobel en 1945, Alexander Fleming ya nos avisó de la habilidad de los microorganismos para adquirir resistencia a antibióticos y del peligro que esto suponía (Fleming, 1945). Desgraciadamente, esta advertencia no ha sido atendida, y el abuso de los antibióticos desde que se introdujeron en la práctica clínica a mediados del siglo pasado ha conducido a un incremento constante de las frecuencias de resistencia en los patógenos bacterianos. De hecho, la diseminación de la resistencia a antibióticos entre patógenos de interés clínico se ha convertido en uno de los principales problemas actuales de salud pública (Figura 1), como reconocen todas las organizaciones internacionales de salud, las Naciones Unidas e incluso el Fondo Monetario Internacional (CDC, 2013; ECDC, 2013; Jonas, Parry, Chisholm, Banatvala y Ladminarayan, 2014; OMS, 2016). Por ejemplo, la Organización Mundial de la Salud subraya que «si no se toman medidas urgentes, el mundo está abocado a una era postantibióticos en la que muchas infecciones comunes y lesiones menores volverán a ser potencialmente mortales» (OMS, 2018). De acuerdo con estimaciones recientes, aproximadamente 700.000 personas mueren cada año en el mundo como consecuencia de infecciones resistentes a los antibióticos (O’Neill, 2016). Estas predicciones también indican que, de no revertirse la tendencia actual de diseminación de la resistencia a antibióticos, para el 2050 las infecciones resistentes podrían convertirse en la primera causa de mortalidad en el mundo y provocar diez millones de muertes anuales.
Adaptándose a los antibióticos
La evolución de la resistencia a antibióticos en las bacterias es un ejemplo simple y elegante de adaptación mediante selección natural (Figura 2). Una bacteria adquiere un mecanismo de resistencia que es seleccionado en presencia del antibiótico. En ausencia del antibiótico, el mecanismo de resistencia generalmente produce un coste biológico en términos de una reducción del fitness o eficacia biológica de la bacteria (definida como una disminución de su tasa reproductiva). Sin embargo, esta reducción de eficacia biológica se puede corregir mediante mutaciones compensatorias que alivian el coste asociado con la resistencia. Considerando esta dinámica, es fácil comprender cómo el uso excesivo de antibióticos ha promovido la diseminación de la resistencia a antibióticos a nivel global.
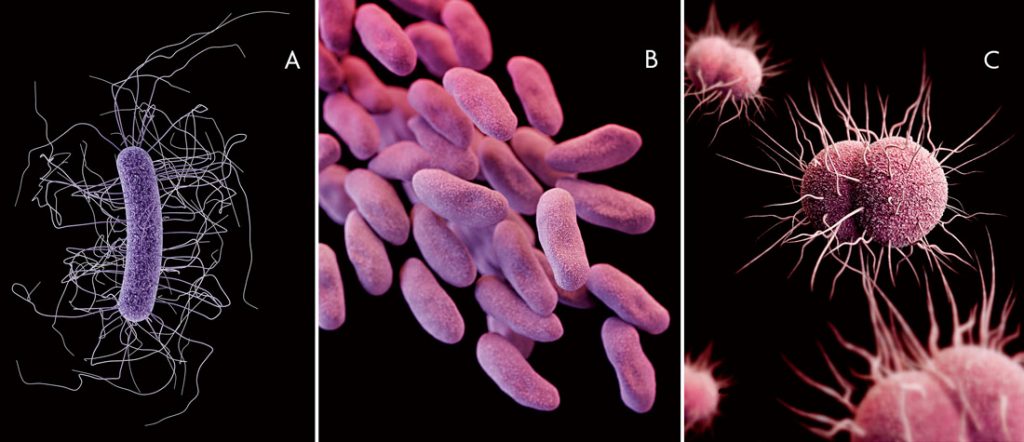
Figura 1. En 2013, el Centro de Control y la Prevención de Enfermedades de Estados Unidos (CDC en sus siglas en inglés) lanzó un informe catalogando las principales amenazas para la salud pública provocadas por la resistencia a antibióticos entre patógenos de interés clínico (CDC, 2013). En la imagen, ilustraciones en 3D de la estructura de las tres bacterias calificadas como «amenaza urgente» en este informe (de izquierda a derecha): A) Clostridium difficile (que puede causar colitis y diarrea de gravedad); B) clones pertenecientes a la familia Enterobacteriaceae resistentes a antibióticos carbapenémicos, y C) cepas resistentes a antibióticos de Neisseria gonorrhoeae (el agente causal de la gonorrea). / CDC / James Archer
La resistencia a antibióticos es, por tanto, un ejemplo fascinante de evolución de innovaciones, ya que una bacteria sensible a un antibiótico necesita innovar para adquirir la resistencia. Lo que es aún más fascinante es el hecho de que para estudiar esta innovación evolutiva no necesitamos investigar restos fósiles, ya que la hemos presenciado en tiempo real, durante las últimas décadas. Es más, podemos incluso reproducir parcialmente este proceso de evolución en nuestros laboratorios, utilizando la llamada «evolución experimental» (Buckling, Craig Maclean, Brockhurst y Colegrave, 2009). Por último, y este es un punto crucial, gracias a los avances tecnológicos, hoy en día podemos determinar exactamente cuáles son las bases genéticas que subyacen a la evolución de la resistencia a antibióticos en las bacterias. En definitiva, todo lo expuesto anteriormente nos sitúa en una posición ideal para responder algunas de las preguntas clave sobre la evolución de innovaciones responsables de la resistencia a antibióticos, y así lo intentaremos en las próximas líneas.
Rutas hacia la resistencia
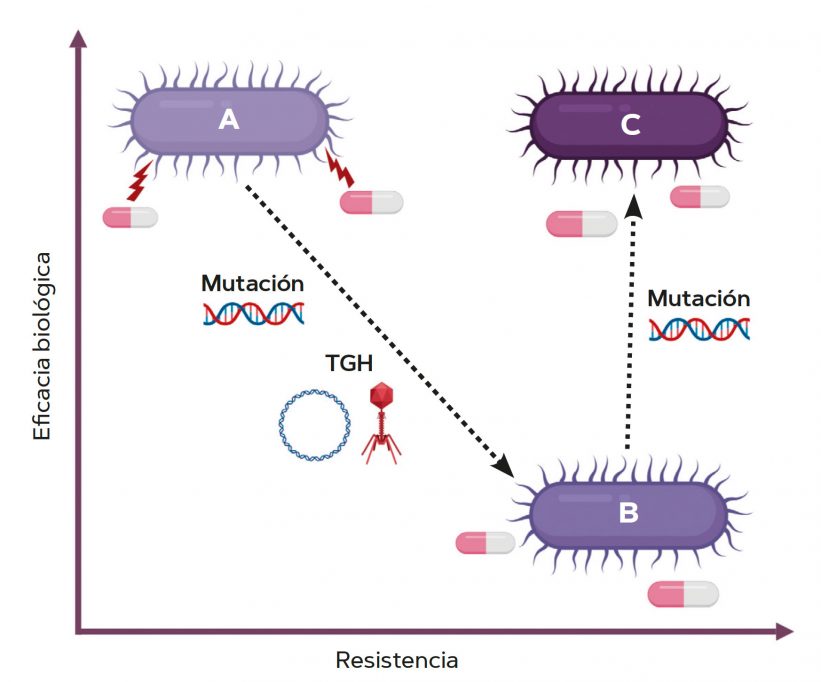
La adquisición de resistencia a antibióticos en las bacterias puede seguir fundamentalmente dos rutas: a) mutaciones cromosómicas y b) la adquisición de mecanismos específicos de resistencia mediante transferencia genética horizontal (Figura 3). Estos dos procesos son fundamentalmente diferentes en varios aspectos clave. Las mutaciones cromosómicas consisten en alteraciones en los genes codificados por la bacteria que desembocan en un fenotipo de resistencia a los antibióticos. Este es un poderoso mecanismo adaptativo, dada la alta tasa de mutación constitutiva de las bacterias (que se ve además incrementada en presencia de algunos antibióticos) y el gran tamaño de las poblaciones bacterianas (que aumenta las probabilidades de que al menos un individuo de la población adquiera la mutación adecuada).
Las mutaciones que confieren resistencia pueden afectar a las dianas celulares de los antibióticos, y modificarlas para que estos no las reconozcan. Por ejemplo, si el mecanismo de acción de un antibiótico se basa en el bloqueo de una enzima bacteriana, las mutaciones que alteren la estructura de esta enzima, y por tanto eviten el reconocimiento por parte del antibiótico (e idealmente sin alterar significativamente su actividad), conducirán a la resistencia. Otro tipo de mutaciones que confieren resistencia son aquellas que producen la disminución de la concentración intracelular de los antibióticos a través de la expulsión o de la reducción de la captación de los mismos. En esta categoría se incluyen, por ejemplo, mutaciones que producen la sobreexpresión de bombas de eflujo que retiran activamente el antibiótico del compartimento intracelular bacteriano. En definitiva, la adquisición de resistencia a través de mutaciones cromosómicas se basa en la alteración del material genético preexistente en la bacteria y se transmiten de forma vertical a la descendencia.

La segunda ruta de adquisición de resistencia a antibióticos es a través de transferencia genética horizontal (TGH). La TGH es el movimiento de material genético entre organismos independiente de la herencia vertical y consiste básicamente en el intercambio directo de ADN entre individuos. Este proceso juega un papel crucial en la evolución de los seres vivos y es particularmente frecuente y relevante en las bacterias, ya que alivia su carencia de reproducción sexual. La TGH en bacterias puede realizarse directamente a través de la captación de ADN desnudo del medio, que posteriormente se recombina con el material genético bacteriano, en un proceso conocido como transformación. Más relevante aún para la evolución de innovaciones en bacterias es la TGH vehiculada por elementos genéticos móviles (EGM), que son estructuras genéticas capaces de transferirse activamente entre bacterias. Entre estos elementos destacan los virus bacteriófagos (o fagos) y los plásmidos, que median los procesos conocidos como transducción y conjugación, respectivamente (Figura 3). Los EGM portan los genes necesarios para su ciclo biológico, pero además codifican múltiples genes accesorios con elevado valor adaptativo para las bacterias. De este modo, los EGM dan acceso a las bacterias receptoras a un repertorio de genes completamente nuevos, de forma que les permite adaptarse a nuevos nichos ecológicos y resistir diferentes insultos ambientales de manera prácticamente inmediata. ¿Imaginan poder adquirir el color de ojos de la persona sentada a su lado, el olfato de su perro o incluso las alas de un pájaro, en cuestión de minutos? Ese es el poder de la TGH en las bacterias.

Figura 4. Además de la investigación en nuevos antibióticos, se hace necesario promover un uso más racional de estos para disminuir la presión selectiva sobre las poblaciones bacterianas. En este sentido, gobiernos de todo el mundo están lanzando campañas de información tanto a la ciudadanía como al personal médico para terminar con el uso abusivo de estas «balas mágicas». En la imagen, póster de campaña del Plan Nacional frente a la Resistencia a los Antibióticos, en activo en España desde 2014 y cuya segunda fase (2019-2021) acaba de iniciarse. / Ministerio de Sanidad, Servicios Sociales e Igualdad. Gobierno de España
Los EGM de mayor relevancia en la evolución de la resistencia a antibióticos en bacterias en los ambientes clínicos son los plásmidos (San Millán, 2018). Los plásmidos son pequeñas moléculas circulares de ADN que se replican independientemente del cromosoma bacteriano y que se transfieren a través de un túnel proteico que establece contacto directo entre bacterias en un proceso conocido como conjugación. Estos elementos genéticos codifican sofisticados mecanismos de resistencia, muchos de ellos adquiridos a partir de las bacterias ambientales que llevan millones de años produciendo los antibióticos que utilizamos actualmente en los hospitales. Y lo que es más preocupante: estos plásmidos portan normalmente no uno, sino varios mecanismos de resistencia diferentes, y confieren así resistencia a múltiples antibióticos en un único evento de conjugación. Además, los plásmidos se mantienen en múltiples copias en las bacterias hospedadoras y proporcionan un aumento en la dosis de los genes que codifican, lo que sirve de combustible para la continua evolución de innovaciones a partir de estos (Rodríguez-Beltrán et al., 2018; San Millán, Escudero, Gifford, Mazel y MacLean, 2016). Por todas estas razones, la resistencia a antibióticos mediada por plásmidos ha emergido como uno de los problemas de más difícil solución en los ambientes clínicos a lo largo de las últimas décadas.
Conclusión
El estudio de la evolución de la resistencia a antibióticos nos está permitiendo comprender algunas de las reglas que gobiernan la evolución de innovaciones en las bacterias, y por extensión en el resto de los seres vivos. La evolución de la resistencia es testimonio de la increíble capacidad adaptativa de las bacterias, que normalmente encuentran no una, sino varias soluciones genéticas para sobrevivir a los antibióticos. Por lo tanto, si queremos seguir disfrutando de los enormes beneficios de estas balas mágicas en el futuro, será necesario promover un uso más racional de los antibióticos que contribuya a disminuir la presión selectiva sobre las poblaciones bacterianas (Figura 4). Finalmente, lo más fascinante del tema objeto de este artículo probablemente sea el hecho de que, durante los minutos que ha tardado en leerlo, todos los mecanismos genéticos de adaptación que describimos han ocurrido en tiempo real en su tracto intestinal, en algunas de las aproximadamente treinta billones bacterias que lo habitan.
REFERENCIAS
Buckling, A., Craig Maclean, R., Brockhurst, M. A., & Colegrave, N. (2009). The Beagle in a bottle. Nature, 457, 824–829. doi: 10.1038/nature07892
CDC. (2013). Antibiotic resistance threats in the United States. Centres for Disease Control and Prevention, US Department of Health and Human Services. Consultado en http://www.cdc.gov/drugresistance/threat-report-2013
ECDC. (2013). Annual epidemiological report 2012. Reporting on 2010 surveillance data and 2011 epidemic intelligence data. Estocolm: ECDC. Consultado en https://ecdc.europa.eu/en/publications-data/annual-epidemiological-report-2012-2010-data
Fleming, A. (1945, 11 de desembre). Penicillin. Nobel Lecture. NobelPrize.org. Consultado en https://www.nobelprize.org/uploads/2018/06/fleming-lecture.pdf
Jonas, O., Parry, I., Chisholm, D., Banatvala, N., & Ladminarayan, R. (2014). Global health threats of the 21st century. Finance and Development, 51(4), 16–20. Consultado en https://www.imf.org/external/pubs/ft/fandd/2014/12/jonas.htm
Mayr, E. (1963). Animal species and evolution. Cambridge, MA: Harvard University Press.
OMS. (2016). United Nations high-level meeting on antimicrobial resistance. Consultado en https://www.who.int/antimicrobial-resistance/events/UNGA-meeting-amr-sept2016/en/
OMS. (2018). Resistencia a los antibióticos. Consultado en https://www.who.int/es/news-room/fact-sheets/detail/resistencia-a-los-antibi%C3%B3ticos
O’Neill, J. (2016). Tackling drug-resistant infections globally: Final report and recommendations. Londres: Review on Antimicrobal Resistance.
Rodríguez-Beltrán, J., Hernández-Beltrán, J. C. R., DelaFuente, J., Escudero, J. A., Fuentes-Hernández, A., MacLean, R. C., … San Millán, A. (2018.) Multicopy plasmids allow bacteria to escape from fitness trade-offs during evolutionary innovation. Nature Ecology & Evolution, 2, 873–881. doi: 10.1038/s41559-018-0529-z
San Millán, A. (2018). Evolution of plasmid-mediated antibiotic resistance in the clinical context. Trends in Microbiology, 26(12), 978–985. doi: 10.1016/j.tim.2018.06.007
San Millán, A., Escudero, J. A., Gifford, D. R., Mazel, D., & MacLean R. C. (2016). Multicopy plasmids potentiate the evolution of antibiotic resistance in bacteria. Nature Ecology & Evolution, 1, 0010. doi: 10.1038/s41559-016-0010
Tan, S. Y., & Grimes, S. (2010). Paul Ehrlich (1854-1915): Man with the magic bullet. Singapore Medical Journal, 51(11), 842–843.
Wagner, A. (2015). Arrival of the fittest: How nature innovates. Nova York: Current.