Y la biología se convirtió en ingeniería
La adopción de estándares para sistemas vivos
Durante décadas, los biólogos moleculares han estado eliminando o insertando genes en todo tipo de organismos con una intención biotecnológica o simplemente para generar conocimiento fundamental. La biología sintética da un paso más allá e incorpora marcos conceptuales procedentes de la computación, la electrónica y el diseño industrial. Este cambio permite plantear la creación de objetos biológicos complejos que anteriormente se consideraban demasiado difíciles de ensamblar. Para ello, hay que adoptar las etapas de cualquier proceso de producción industrial: diseño, fabricación de los componentes, montaje y manufactura final. Este objetivo hace necesario estandarizar los formatos físicos y funcionales de los componentes implicados, los métodos de ensamblaje de ADN, las medidas de actividad y los lenguajes descriptivos.
Palabras clave: biología sintética, estándares, represilador, repositorio, ortogonalidad.
El efecto fundacional: la biología vista por los ingenieros
Aunque la historia de la biología sintética que conocemos se remonta mucho tiempo atrás, el nacimiento de su versión contemporánea puede ubicarse claramente en el Instituto Tecnológico de Massachusetts (MIT, en sus siglas en inglés) al principio de los años 2000. En esa época, Tom Knight, un profesor de inteligencia artificial en el Departamento de Ciencias de la Computación del MIT, comenzó a formalizar la idea de abordar los sistemas biológicos utilizando como marco interpretativo toda la artillería conceptual de la ingeniería eléctrica, industrial y computacional. Para ello, era preciso adaptar a las entidades vivas las abstracciones fundamentales de los ingenieros cuando analizan y diseñan objetos con distintos grados de complejidad. De esta manera, el conocido como dogma central de la biología molecular se sustituye por un marco interpretativo altamente abstracto en el que las partes biológicas dan lugar a los dispositivos y estos, a los módulos y sistemas (Figura 1). Y el contexto evolutivo para explicar el origen de las funciones biológicas se aparca para poner todo el énfasis en la lógica relacional que hace que los sistemas vivos funcionen aquí y ahora (De Lorenzo, 2018).
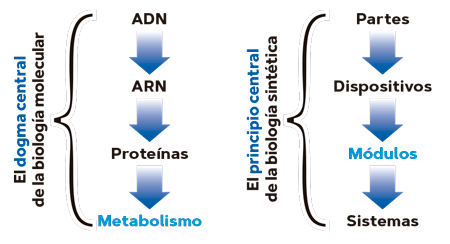
Figura 1. Del dogma central de la biología molecular al marco conceptual de la biología sintética. En el primer caso, el énfasis es la transferencia de información del ADN a las proteínas y el metabolismo. En contraste, la biología sintética se preocupa de la lógica relacional y composicional de los sistemas vivos y aplica abstracciones típicas de la ingeniería a los objetos biológicos. / Fuente: De Lorenzo (2018)
Esta «opción hermenéutica» permite una visión de los sistemas vivos compatible pero muy distinta a la de la biología molecular. Mientras que esta –fundada por científicos atómicos de la posguerra mundial– promovió una visión de las entidades biológicas a través de los ojos de la física, la biología sintética ambiciona reinterpretarlas a través de la ingeniería (Andrianantoandro, Basu, Karig y Weiss, 2006). Un efecto colateral de este nuevo punto de vista es que, a diferencia de la biología molecular, la agenda de la biología sintética no tiene que ver solo con la comprensión de los objetos biológicos existentes, sino también con su modificación racional para dar lugar a propiedades y funcionalidades nuevas, en algunos casos, de alto valor económico. En realidad, uno de los grandes horizontes de la biología sintética es hacer rigurosamente real la palabra ingeniería que de forma metafórica se asoció a genética durante la revolución del ADN recombinante a finales de los años setenta del pasado siglo. Por el contrario, el discurso fundacional de la biología sintética declara que es posible pasar de la analogía a la metodología y del bricolage genético de prueba y error al planteamiento racional de sistemas vivos complejos tal y como un ingeniero hace con sus diseños técnicos (De Lorenzo, 2018; Endy, 2005). Para ello, es preciso primero deconstruir los sistemas biológicos existentes en un catálogo de partes que, una vez estandarizadas, pueden reconectarse con una lógica predeterminada para generar funcionalidades nuevas.
«La biología sintética ambiciona la reinterpretación de las entidades biológicas a través de la ingeniería»
Del triunfo de este escenario da cuenta el extraordinario éxito del llamado Repositorio de Partes Biológicas Estandarizadas inicialmente creado y depositado en el MIT y que creció en paralelo a la competición internacional de máquinas genéticas o iGEM, también promovida en su día por los teóricos de la biología sintética en la misma institución (Galdzicki, Rodriguez, Chandran, Sauro y Gennari, 2011). Esta competición, que llega hasta el día de hoy, ha sido fundamental para propagar la idea de la biología-como-ingeniería a través de numerosas universidades en todo el mundo y ha sido en sí misma un inmenso experimento educacional que ha iniciado a varias generaciones de jóvenes en los conceptos fundamentales de la biología sintética.
Pero retrocedamos un momento al principio. ¿En qué se basaba el optimismo manifestado en los comienzos por Tom Knight y sus colegas del MIT? En los años previos a la conceptualización de la biología sintética, aparecieron tres artículos que muchos consideran fundacionales del campo, aunque en ninguno de los casos así se exprese explícitamente en el texto de las publicaciones correspondientes. Uno de ellos fue la descripción en el año 2000 por el grupo de M. Elowitz del llamado represilador: un circuito genético entre tres represores transcripcionales mutuamente inhibidores que en ciertas condiciones dan lugar a la expresión cíclica de un gen reportero (que, en este caso, codificaba una proteína fluorescente), que actúa como indicador visual del funcionamiento del sistema (Elowitz y Leibler, 2000; Figura 2A). El ángulo revolucionario de este trabajo era que ese comportamiento estaba diseñado racionalmente con principios de ingeniería y con una configuración totalmente artificial (no hay casos similares en el mundo natural). Aún así, el comportamiento respondía fielmente a un modelo matemático simulable en un ordenador.
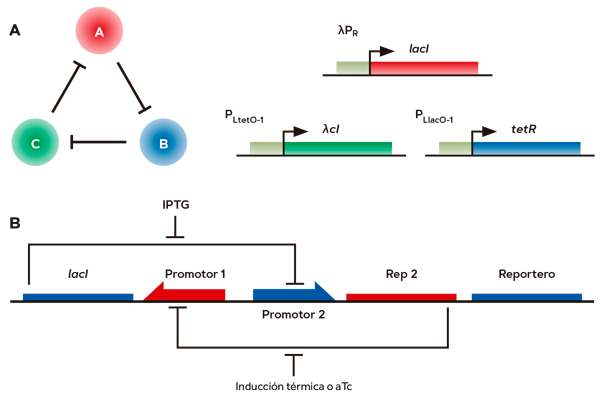
Figura 2. Dos de las construcciones genéticas fundadoras de la biología sintética. A) El represilador. Este dispositivo genético es un oscilador de la expresión genética in vivo que se construye con tres represores, las proteínas LacI, TetR, λCI (codificadas por los genes lacI, tetR, λcI), y donde cada una actúa como inhibidora de las siguientes a base de reprimir los promotores correspondientes (λPR, PLlacO-1 y PLtetO-1), cada uno de los cuales controla la expresión del represor siguiente. Este comportamiento estaba diseñado con principios de ingeniería y con una configuración totalmente artificial (no hay casos similares en el mundo natural). El comportamiento respondía fielmente a un modelo matemático simulable en un ordenador. B) El interruptor. Este sistema se basa en dos de los represores transcripcionales usados en el represilador, pero organizados de forma que se inhiben mutuamente (LacI inhibe al promotor de tetR y viceversa). Esto origina dos estados estables en los que se activa un promotor o se activa el otro, nunca los dos al mismo tiempo. Esto puede revertirse con la adición de una señal inductora (por ejemplo, mediante el uso del reactivo IPTG, o de la anhidrotetraciclina o aTc), que desreprime uno de los componentes. A su vez, esto permite la expresión del otro represor y provoca un cambio de estado del dispositivo. De nuevo, el sistema in vivo obedecía escrupulosamente las reglas impuestas por su diseñador humano y respondía al modelo predictivo. / Fuente: Figura A: Elowitz y Leibler (2000), y Figura B: Gardner, Cantor y Collins (2000)
El segundo artículo fundacional fue publicado por el grupo de J. Collins en el mismo año y describía el diseño y funcionamiento de un interruptor genético (toggle switch), ensamblado con dos de las partes biológicas (los represores transcripcionales) utilizadas en el caso anterior (Gardner, Cantor y Collins, 2000; Figura 2B). Pero, en este caso, cableadas de una forma muy distinta y que daba lugar a la expresión estable de dos genes alternativos. De nuevo, el sistema in vivo obedecía escrupulosamente las reglas impuestas por su diseñador humano y respondía al modelo predictivo. En una tercera publicación en 2000 del laboratorio de L. Serrano, se describían circuitos genéticos simples en los que se introducían de forma racional lazos de retroalimentación negativos que daban estabilidad al sistema (Becskei y Serrano, 2000). En cada uno de los tres casos el mensaje era el siguiente: se pueden aplicar principios de ingeniería a los sistemas vivos, tanto para entender cómo funcionan como para hacer que funcionen predeciblemente de otra manera. El impacto conceptual de estas publicaciones fue enorme. Y tras la correspondiente formulación de la disciplina en el MIT por Knight y sus colaboradores Drew Endy (Endy, 2005) y Ron Weiss (Andrianantoandro et al., 2006) podemos decir que la biología sintética había nacido.
El abecé de la biología sintética
La noción básica que hay tras la biología sintética es que cualquier sistema biológico puede considerarse como una combinación compleja de elementos funcionales e independientes, no muy diferentes a los que se encuentran en los dispositivos hechos por el hombre. Sobre esta premisa, los objetos correspondientes pueden ser descritos como la combinación de un número limitado de componentes y reconstruidos en una configuración diferente, bien para modificar las propiedades existentes o para conseguir otras completamente nuevas. Un aspecto fundamental de la biología sintética es el desarrollo de herramientas materiales y conceptuales de uso general (partes biológicas, genomas mínimos, células artificiales, síntesis de ADN…) para abordar problemas hasta ahora irresolubles como la biosíntesis de moléculas complejas, la descomposición o reciclaje de productos químicos tóxicos, la detección biológica de explosivos, la producción biológica de H2 y otros combustibles, etc. (De Lorenzo et al., 2018). Pero estas mismas técnicas permiten también abordar desafíos completamente novedosos, como la computación con ADN, el diseño de patrones de desarrollo, el uso de bacterias para eliminar tumores, la expansión del código genético a aminoácidos no naturales y otras muchas aplicaciones sorprendentes (De Lorenzo et al., 2018; O’Day et al., 2019).
«El comportamiento de componentes biológicos independientemente de su contexto es un requisito previo para la ingeniería de nuevos dispositivos y propiedades»
Una premisa recurrente en biología sintética es la necesidad de estandarizar los componentes biológicos de una forma separada a la que tienen en sus circunstancias naturales. El comportamiento de esos componentes independientemente de su contexto es un requisito previo para la ingeniería de nuevos dispositivos y propiedades. Si bien la necesidad y la oportunidad de tal estandarización han sido identificadas claramente, el éxito del esfuerzo ha sido bastante limitado hasta ahora. A pesar de la larga lista de partes biológicas depositadas en el registro respaldado por el MIT, todavía hay un largo camino para cumplir los requisitos que las elevarían al nivel de los estándares utilizados por la industria civil o electrónica (De Lorenzo y Schmidt, 2018). Esto se debe en parte a la propiedad intrínseca de las funciones biológicas de evolucionar conjuntamente como un todo y, por lo tanto, comportarse de una manera muy dependiente del contexto. Por lo tanto, existe la necesidad de desarrollar mejores conceptos y un lenguaje especial para tratar y clasificar tales partes biológicas, que se base no solo en su posible similitud con sus equivalentes electrónicos, sino también en una mejor comprensión de las funciones biológicas mínimas, principalmente relacionadas con la regulación de la expresión génica (De Lorenzo y Danchin, 2008). Estas mejoras pueden sentar las bases de un futuro acuerdo internacional sobre el formato de dichas partes, su disponibilidad y el registro de sus usuarios.
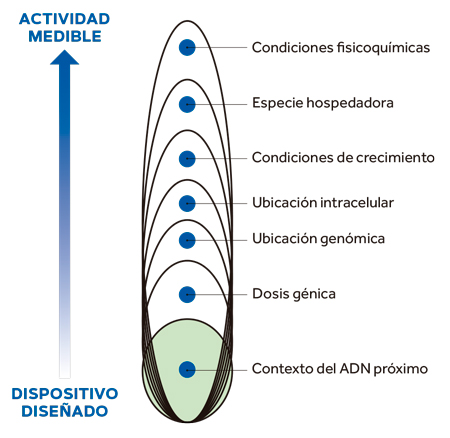
Figura 3. Niveles contextuales de cualquier construcción genética. La figura resume cómo la funcionalidad de una construcción tiene que pasar por varias capas de contextos biológicos y fisicoquímicos antes de que el observador pueda medir su actividad en situaciones diferentes y comprobar así su potencial estandarización. / Fuente: Porcar, Danchin y De Lorenzo (2014)
Como se muestra en la Figura 3, el funcionamiento de cada dispositivo de expresión en una célula está sujeto al menos a siete capas contextuales que van desde la influencia mutua inmediata de las secuencias de ADN adyacentes hasta las condiciones fisicoquímicas ambientales. Es esencial racionalizar este itinerario desde la composición física hasta la función final, y de una forma medible y cuantificable. Esto exige tres abordajes convergentes. Primero, el modelado detallado, la medición y la parametrización de grandes colecciones de partes funcionales en una variedad de contextos de ADN y condiciones de crecimiento. En segundo lugar, la investigación de un número limitado de promotores arquetípicos (es decir, basados en secuencias de ADN consenso o muy bien caracterizados) en diversos entornos genómicos y celulares para identificar y, en última instancia, eliminar los determinantes de la dependencia del contexto. Y tercero, el diseño avanzado de dispositivos de expresión ortogonales (es decir, independientes del contexto genético y fisiológico) posiblemente basados en partes biológicas reclutadas de elementos genéticos móviles (Rao, 2012). El tema de los ribosomas ortogonales y los códigos genéticos alternativos son áreas de investigación fascinantes que podrían también ser objeto de un esfuerzo de estandarización (Wang, Neumann, Peak-Chew y Chin, 2007).
Estándares α y β para hacer los sistemas vivos más fáciles de diseñar
En el mundo de la ingeniería, los términos estándar y estandarización se refieren principalmente a tres cuestiones. En primer lugar, a la adopción de formas geométricas específicas y formatos de tamaño para el ensamblaje físico de los componentes de un sistema hecho por el hombre. También hacen referencia a la definición de unidades de medida de propiedades y parámetros relevantes, así como a las condiciones y procedimientos para calcularlos (por ejemplo, amperios para corriente, ohmios para resistencia, etc.). Y, por último, a la implementación de protocolos inequívocos para la fabricación de objetos. Estos estándares permiten la abstracción de las propiedades de los componentes de un sistema, su descripción precisa con un lenguaje cuantitativo adecuado, también estandarizado, y la modelización del objeto diseñado con idénticos métodos de representación. Una gran ventaja a este respecto es la posibilidad de disociar el diseño detallado de un producto de la fabricación de sus componentes y el ensamblaje final del artefacto. Esto es común en la ingeniería industrial y electrónica, pero, ¿cuánto de esto puede importarse al ámbito biológico?
«La biología molecular y la biotecnología se han visto afectadas desde su nacimiento por un desprecio casi total por el reto de la estandarización»
La biología molecular y la biotecnología se han visto afectadas desde su nacimiento por un desprecio casi total por el reto de la estandarización. Existe una nomenclatura generalmente caótica para genes y herramientas moleculares. La ausencia de estándares ha dificultado la medida comparativa de funciones biológicas tan básicas como la fuerza de un promotor (Beal et al., 2016; Popp, Dotzler, Radeck, Bartels y Mascher, 2017). Por lo tanto, es importante examinar cuáles de estas funciones pueden ser objeto de un esfuerzo de estandarización que incluso podría derivar en una acción prenormativa. Pero, ¿qué se puede estandarizar con el nivel de conocimiento que tenemos en este momento?
Podríamos distinguir entre estándares en versión β y α. El primero es el conjunto de reglas que adopta una cierta comunidad para mejorar la comunicación, la cooperación y la interoperabilidad, pero sin pretensiones de universalidad. El punto de partida más común es el establecimiento de cánones para la composición física de dispositivos biológicos. Hoy en día existen múltiples formatos que permiten un fácil ensamblaje de partes de ADN y módulos individuales para generar sistemas más complejos (Casini, Storch, Baldwin y Ellis, 2015). Otro ejemplo es la introducción de microesferas fluorescentes para calibrar y comparar los experimentos de citometría (es decir, el contaje y la media de las emisiones ópticas de células individuales en una población), como se ha propuesto recientemente (Beal et al., 2019). Estos estándares son necesariamente transitorios, porque en algún momento el abaratamiento de la síntesis de ADN o la aparición de nuevas plataformas de medida de la fluorescencia los hará innecesarios.
«La adopción de formalismos derivados de la ingeniería eléctrica e industrial ha sido extraordinariamente útil para el desarrollo de la biología sintética»
Pero hay un segundo tipo de estándares (los que he llamado α) especialmente relacionados con la metrología de las actividades biológicas que deberían de desarrollarse, implementarse y finalmente promulgarse. En mi opinión, los más importantes están relacionados con la medida del flujo de expresión génica desde una secuencia codificante de ADN hasta una proteína. Esto está aún por desarrollar conceptualmente y materialmente. Las unidades de ARN polimerasa por segundo (PoPS) y de ribosoma por segundo (RiPS) como referencia universal para expresar la fuerza del promotor de la maquinaria de traducción son necesarias, pero estas unidades no se han abordado seriamente ni desde un punto de vista fundamental ni como una tecnología de medida (Kelly et al., 2009; Sendy, Lee, Busby y Bryant, 2016). Algunos argumentan que la inteligencia artificial y el diseño asistido por ordenador (CAD) serán capaces de anticipar la mayoría de los escenarios posibles dentro de un proyecto de expresión de cualquier gen de interés a base de compilar un gran número de datos experimentales sobre transcripción, degradación y traducción de ARN mensajeros (Kosuri et al., 2013). Pero ¿será esto alcanzable a corto plazo? La traducción es en sí misma una función compleja que depende no solo de la secuencia del ARN mensajero, sino también de su estabilidad y de un gran número de parámetros fisiológicos como la fase de crecimiento, el estrés ambiental o la distribución de recursos celulares. La medida de la velocidad de plegamiento y las modificaciones postraduccionales son retos difícilmente abordables aún. Quizá, como es el caso en otras ramas de la ingeniería, los diseños biológicos del futuro dependerán por completo del aprendizaje automático basado en la experiencia (Salis, Mirsky y Voigt, 2009). Pero entre tanto, el diseño racional de sistemas vivos estará limitado por la adopción de estándares tanto β como α.

Figura 4. Los estándares necesitan objetos materiales de referencia. A) Patrón del metro. Durante la Revolución francesa, la Convención Nacional mandó instalar muestras de la nueva medida en mármol blanco por los distintos barrios de París. En la imagen, patrón instalado en la calle de Vaugirard. B) Patrón del kilo. Esta medida es otro producto de la Revolución francesa. Debido a que su definición como el peso de un litro de agua no era precisa, se redefinió usando como referencia un objeto cilíndrico fabricado con platino e iridio que se guarda en la Oficina Internacional de Pesas y Medidas en París. El kilo ha sido redefinido recientemente en términos absolutos a través de la mecánica cuántica. En la imagen, réplica del kilo del Instituto Nacional de Normas y Tecnología de Estados Unidos, situado en Gaithersburg, en el estado de Maryland. / Foto izquierda: LPTP / Wikimedia Commons; foto derecha: National Institute of Standards and Technology Digital Collections, Gaithersburg, MD 20899
¿Qué hacer?
«Para describir, medir y “recablear” las funcionalidades biológicas rigurosamente, e intercambiar datos, es necesario adoptar estándares»
Es cierto que los sistemas biológicos no pueden equipararse automáticamente a los artefactos humanos. Pero la adopción de formalismos derivados de la ingeniería eléctrica e industrial ha sido extraordinariamente útil para el desarrollo de la biología sintética. La progresión de partes a dispositivos y de estos a módulos y sistemas es ahora un marco conceptual perfectamente aceptado en cualquier proyecto de biología sintética. Pero para describir, medir y «recablear» las funcionalidades biológicas rigurosamente, e intercambiar datos, es necesario adoptar estándares (β en el corto plazo y α de forma duradera). Más allá de las reglas para la composición física de las secuencias de ADN, se necesita urgentemente el desarrollo de un nuevo tipo de tecnologías que podríamos designar como metrología biomolecular in vivo. El objetivo no es solo proponer unidades inequívocas para describir las correspondientes actividades (transcripción, traducción, etc.) sino también generar entidades materiales para una calibración de referencia con el fin de permitir la coordinación de mediciones en distintos tiempos y lugares. Es interesante que en la historia de los estándares se haya recurrido a objetos materiales (el kilo, el metro, etc.) cuidadosamente guardados como referencia de muchos tipos de medidas (Figura 4). ¿Se podrá ir por el mismo camino en el mundo biológico?
Referencias
Andrianantoandro, E., Basu, S., Karig, D. K., & Weiss, R. (2006). Synthetic biology: New engineering rules for an emerging discipline. Molecular Systems Biology, 2(1), 2006.0028. doi: 10.1038/msb4100073
Beal, J., Farny, N. G., Haddock-Angelli, T., Selvarajah, V., Baldwin, G. S., Buckley-Taylor, R., … Workman, C. T. (2019). Robust estimation of bacterial cell count from optical density. BioRxiv, 803239. doi: 10.1101/803239
Beal, J., Haddock-Angelli, T., Gershater, M., De Mora, K., Lizarazo, M., Hollenhorst, J., & Rettberg, R. (2016). Reproducibility of fluorescent expression from engineered biological constructs in E. coli. PLOS ONE, 11(3), e0150182. doi: 10.1371/journal.pone.0150182
Becskei, A., & Serrano, L. (2000). Engineering stability in gene networks by autoregulation. Nature, 405(6786), 590. doi: 10.1038/35014651
Casini, A., Storch, M., Baldwin, G. S., & Ellis, T. (2015). Bricks and blueprints: Methods and standards for DNA assembly. Nature Reviews Molecular Cell Biology, 16(9), 568–576. doi: 10.1038/nrm4014
De Lorenzo, V. (2018). Evolutionary tinkering vs. rational engineering in the times of synthetic biology. Life Sciences, Society and Policy, 14(18). doi: 10.1186/s40504-018-0086-x
De Lorenzo, V., & Danchin, A. (2008). Synthetic biology: Discovering new worlds and new words. EMBO Reports, 9(9), 822–827. doi: 10.1038/embor.2008.159
De Lorenzo, V., & Schmidt, M. (2018). Biological standards for the Knowledge-Based BioEconomy: What is at stake. New Biotechnology, 40, 170–180. doi: 10.1016/j.nbt.2017.05.001
De Lorenzo, V., Prather, K. L., Chen, G. Q., O’Day, E., Von Kameke, C., Oyarzun, D. A., ... Lee, S. Y. (2018). The power of synthetic biology for bioproduction, remediation and pollution control: The UN’s Sustainable Development Goals will inevitably require the application of molecular biology and biotechnology on a global scale. EMBO Reports, 19(4), e4658. doi: 10.15252/embr.201745658
Elowitz, M. B., & Leibler, S. (2000). A synthetic oscillatory network of transcriptional regulators. Nature, 403(6767), 335–338. doi: 10.1038/35002125
Endy, D. (2005). Foundations for engineering biology. Nature, 438(7067), 449–453. doi: 10.1038/nature04342
Galdzicki, M., Rodriguez, C., Chandran, D., Sauro, H. M., & Gennari, J. H. (2011). Standard biological parts knowledgebase. PLoS ONE, 6(2), e17005. doi: 10.1371/journal.pone.0017005
Gardner, T. S., Cantor, C. R., & Collins, J. J. (2000). Construction of a genetic toggle switch in Escherichia coli. Nature, 403(6767), 339–342. doi: 10.1038/35002131
Kelly, J. R., Rubin, A. J., Davis, J. H., Ajo-Franklin, C. M., Cumbers, J., Czar, M. J., ... Endy, D. (2009). Measuring the activity of BioBrick promoters using an in vivo reference standard. Journal of Biological Engineering, 3(1), 4. doi: 10.1186/1754-1611-3-4
Kosuri, S., Goodman, D. B., Cambray, G., Mutalik, V. K., Gao, Y., Arkin, A. P., ... Church, G. M. (2013). Composability of regulatory sequences controlling transcription and translation in Escherichia coli. Proceedings of the National Academy of Sciences, 110(34), 14024–14029. doi: 10.1073/pnas.1301301110
O’Day, E., Hosta-Rigau, L., Oyarzún, D. A., Okano, H., De Lorenzo, V., Von Kameke, C., ... Lee, S. Y. (2019). Are we there yet? How and when specific biotechnologies will improve human health. Biotechnology Journal, 14(1), e1800195. doi: 10.1002/biot.201800195
Popp, P. F., Dotzler, M., Radeck, J., Bartels, J., & Mascher, T. (2017). The Bacillus BioBrick Box 2.0: Expanding the genetic toolbox for the standardized work with Bacillus subtilis. Scientific Reports, 7(1), 15058. doi: 10.1038/s41598-017-15107-z
Porcar, M., Danchin, A., & De Lorenzo, V. (2014). Confidence, tolerance, and allowance in biological engineering: The nuts and bolts of living things. Bioessays, 37(1), 95, doi: 10.1002/bies.201400091
Rao, C. V. (2012). Expanding the synthetic biology toolbox: Engineering orthogonal regulators of gene expression. Current Opinion in Biotechnology, 23(5), 689–694. doi: 10.1016/j.copbio.2011.12.015
Salis, H. M., Mirsky, E. A., & Voigt, C. A. (2009). Automated design of synthetic ribosome binding sites to control protein expression. Nature Biotechnology, 27(10), 946–950. doi: 10.1038/nbt.1568
Sendy, B., Lee, D. J., Busby, S. J., & Bryant, J. A. (2016). RNA polymerase supply and flux through the lac operon in Escherichia coli. Philosophical Transactions of the Royal Society B: Biological Sciences, 371(1707), 20160080. doi: 10.1098/rstb.2016.0080
Wang, K., Neumann, H., Peak-Chew, S. Y., & Chin, J. W. (2007). Evolved orthogonal ribosomes enhance the efficiency of synthetic genetic code expansion. Nature Biotechnology, 25(7), 770–777. doi: 10.1038/nbt1314