Plantas a la carta
La edición de genomas para la mejora vegetal

Las plantas que comemos son el resultado de un largo proceso de domesticación de especies silvestres realizado por los seres humanos. En la actualidad la tecnología de edición de genes CRISPR/Cas proporciona una nueva aproximación para la mejora de los cultivos a la vez que ofrece un interesante espectro de posibilidades en la obtención de variedades con características nuevas y más saludables. La tecnología se apoya en dos pilares fundamentales: por un lado, el conocimiento de la secuencia de genomas completos y, por otro, la identificación de la función de los genes. En menos de una década, diseñar plantas a la carta ha pasado de ser una utopía a una posibilidad real.
Palabras clave: cultivos, mejora genética, CRISPR/Cas9, edición de genes.
Las plantas que comemos
Las plantas son componentes esenciales de los ecosistemas terrestres e imprescindibles para el mantenimiento de la vida en la Tierra. El reino Plantae incluye una enorme diversidad de grupos: briófitas (musgos y hepáticas), pteridofitas (helechos y equisetos), gimnospermas y el extenso grupo de las plantas con flores o angiospermas (monocotiledóneas y dicotiledóneas). La mayor diversidad de especies, y posiblemente el mayor número de especies todavía sin identificar, se concentra en las zonas tropicales del planeta.
«A pesar de la aparente variedad de plantas utilizadas, únicamente en torno a 200 se consumen en todo el mundo de manera habitual»
Se estima que existen entre 450.000 y 500.000 especies diferentes de plantas,1 de las cuales más de la mitad son comestibles. A pesar de la aparente variedad de plantas que se utilizan en la alimentación humana, únicamente en torno a 200 se consumen en todo el mundo de manera habitual. Además, tres plantas –maíz, arroz y trigo– aportan casi un 60 % de las proteínas y calorías de origen vegetal en la dieta de las personas (FAO, 1999). La pérdida de agrobiodiversidad vegetal es una tendencia que ha ido en aumento en los últimos cien años y que está asociada a los cambios producidos en las prácticas de cultivo y a la globalización de los sistemas de producción. El resultado es el reemplazo de múltiples variedades locales por un número reducido de nuevas variedades mejoradas o por variedades exóticas.
Los retos actuales de la agricultura pasan por erradicar el hambre y la malnutrición y asegurar alimentos suficientes para dar respuesta a la demanda de las generaciones presentes y futuras (FAO, 1983). En las estimaciones de la inseguridad alimentaria en el mundo llevadas a cabo por la FAO en 2012, se aprecia que el crecimiento económico es particularmente eficaz para reducir el hambre y la malnutrición. Por otra parte, el crecimiento económico debe incluir la dimensión de la nutrición incidiendo en la diversificación de la dieta y en la adecuada ingesta de nutrientes dentro de sistemas de producción y consumo sostenibles (FAO, FIDA y PMA, 2012).
En años recientes, la prevalencia de las alergias alimentarias y sus efectos globales en la población mundial han sido objeto de análisis. Las sensibilidades a alimentos pueden clasificarse como alergias si implican reacciones inmunológicas anormales a componentes del alimento o como intolerancias si el individuo presenta sensibilidad a alimentos en ausencia de respuesta inmunológica. La sensibilidad a productos de origen vegetal incluye algunos de los alimentos básicos en la dieta humana, como los cereales con gluten (trigo, cebada, centeno, avena, espelta y sus variedades híbridas o subproductos), la soja y frutos secos, como los cacahuetes y las nueces. En particular la intolerancia al gluten afecta al 1 % de la población europea. El tratamiento recomendado para las personas alérgicas o intolerantes es la eliminación de la dieta del alimento o aditivo que provoca la reacción adversa. En este sentido, la industria alimentaria debe proporcionar la mayor cantidad de información en el etiquetado de sus productos (Taylor y Hefle, 2001).
Innovación en mejora vegetal
La mejora de las especies vegetales para consumo es una práctica ancestral que ha tenido como resultado la generación de plantas que presentan nuevos caracteres (Figura 1). Se ha utilizado para obtener plantas de mayor calidad, con mayor rendimiento o más resistentes a condiciones ambientales adversas y a patógenos. Además, la mejora vegetal también se ha dirigido a obtener variedades de plantas con caracteres totalmente nuevos (color y morfología de la planta o el fruto, ausencia de semillas, etc.), no presentes en la especie original. Las técnicas convencionales de mejora vegetal obtienen su mayor fuente de variabilidad genéticas de las mutaciones que se pueden originar de manera espontánea durante los procesos de mitosis o meiosis o mediante mutagénesis provocada (utilizando compuestos químicos o irradiación).
«La mejora de las especies vegetales es una práctica ancestral que ha tenido como resultado la generación de plantas con nuevos caracteres»
Frecuentemente la mutación asociada al carácter deseado debe incorporarse en otra planta mediante hibridación y posterior selección en la población resultante del cruce. De esta manera se transfieren uno o pocos genes desde una variedad o especie donante a otra variedad que actúa como receptor (parental recurrente). Se requieren varias generaciones en las que se vuelve a cruzar la descendencia con el parental recurrente y se selecciona la presencia del carácter que queremos introducir. Este proceso tiene limitaciones en cuanto a la precisión de los fragmentos del genoma introducidos en el recurrente y requiere largos periodos de tiempo para introducir el carácter deseado. En los últimos años, el desarrollo de los métodos de secuenciación masiva ha permitido conocer los genomas completos de muchos cultivos. Esta información permite combinar las metodologías de mejora convencional con herramientas moleculares y acelerar los procesos de selección en los materiales generados.
La aparición de las técnicas de ingeniería genética supuso un salto sustancial en la capacidad para generar nuevas variedades de plantas genéticamente modificadas o GMO (organismos genéticamente modificados). Durante los últimos treinta años el trabajo de numerosos laboratorios ha dado lugar al desarrollo de una gran variedad de plantas con modificaciones de interés; sin embargo, aunque su implantación para uso comercial ha sido muy importante, se reduce a una docena de cultivos diferentes (Beltrán, 2018). Las dos principales líneas de mejora de cultivos se han centrado en la obtención de plantas tolerantes a herbicidas y plantas resistentes a insectos.
Una de las principales causas del bajo número de variedades modificadas presentes en el mercado es el elevado costo de los procesos de obtención de permisos para la autorización de nuevas variedades. Por otra parte, la percepción social, principalmente en Europa, de que los alimentos transgénicos pueden suponer un riesgo para la salud ha frenado su utilización y la aparición de nuevas variedades. Sin embargo, una exhaustiva revisión de estudios científicos publicados durante estos años no ha encontrado ninguna evidencia de que las variedades obtenidas mediante ingeniería genética supongan un riesgo para la salud o para el medio ambiente mayor que las variedades obtenidas por mejora convencional (National Academies of Sciences & Medicine, 2016).
La edición de genes en plantas de cultivo: innovación y mejora
Desde hace escasamente seis años, una nueva tecnología permite la edición precisa de genes. La tecnología CRISPR/Cas2 (CRISPR-associated) hace posible la mejora de variedades vegetales sin la introducción de elementos foráneos. En particular, el sistema CRISPR/Cas9 por su simplicidad (Figura 2), bajo coste y aplicabilidad a un extenso número de especies, está demostrando su enorme potencial.
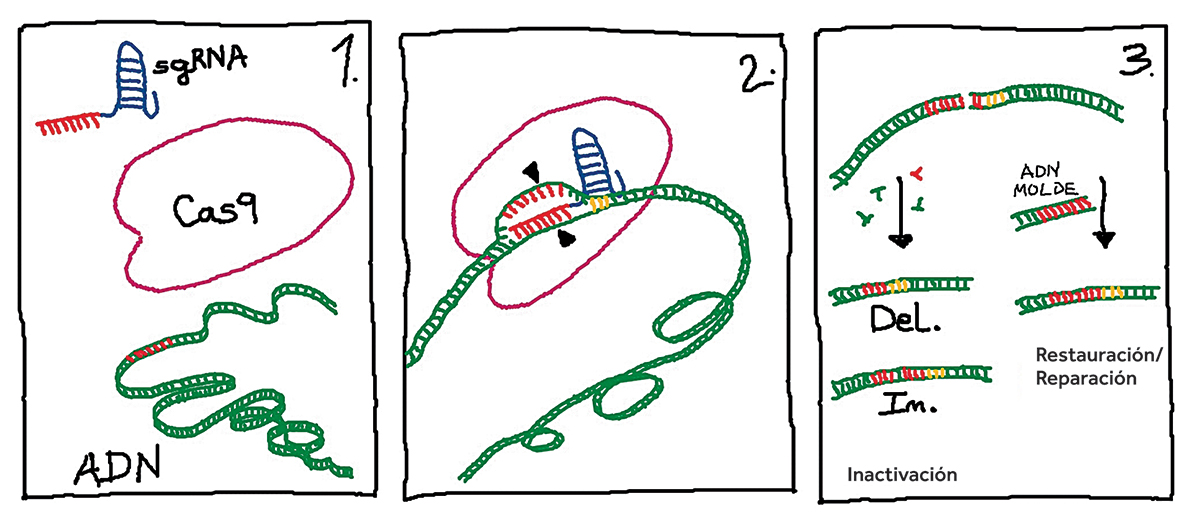
Figura 2. Etapas básicas de la edición de genes mediante la tecnología CRISPR/Cas9. En la etapa 1, los elementos de edición (sgRNA y proteína Cas9) se introducen en las células a editar donde se encuentra el material genómico (ADN). El sgRNA lleva una secuencia de 20 nucleótidos o secuencia guía (en color rojo) complementaria al gen diana. En la etapa 2, el complejo sgRNA/Cas9 interacciona con el ADN genómico y se produce el apareamiento entre la secuencia guía del sgRNA y el gen diana. La proteína Cas9 reconoce una secuencia de tres nucleótidos denominada PAM (en color amarillo) y realiza un corte en la doble hebra de ADN. En la etapa 3, la maquinaria celular activa los mecanismos celulares de reparación. En el lado izquierdo la reparación del corte puede producirse a expensas de introducirse (In.: inserciones) o perderse (Del.: supresiones) nucleótidos, lo cual generalmente lleva a la inactivación del gen. En el lado derecho se muestra el mecanismo de reparación por recombinación homóloga en el que se requiere un ADN molde que permite restaurar la secuencia original o introducir un cambio de nuestra elección. / Concha Gómez Mena
El descubrimiento de los sistemas CRISPR/Cas se remonta a los estudios de sistemas de inmunidad adaptativa microbiana (Mojica y Montoliu, 2016). Algunos microorganismos, principalmente arqueas, se protegen frente a los virus cortando su material genético. Fragmentos de este material genético externo se mantienen en el genoma bacteriano como un recuerdo de la invasión y, en caso de una nueva infección, guiarán a las nucleasas (proteínas Cas) para su corte e inactivación. Eric Lander (2016) publicó un detallado resumen de los principales investigadores implicados en el descubrimiento y estudio del mecanismo molecular de este sistema.
El desarrollo de la tecnología CRISPR/Cas9 para la edición de genes en eucariotas se basa en la reconstitución in vitro de tres de los elementos identificados en Streptococcus pyogenes, os ARN (crRNA y tracrRNA) y la proteína Cas9. Los crRNA (CRISPR RNA) contienen las secuencias homólogas a genes diana presentes en el genoma a editar, mientras que los tracrRNA sirven de anclaje a la proteína Cas9. La proteína Cas9 es una endonucleasa que ocasiona cortes en la doble hebra de ADN. El único requisito para el corte es la presencia de una secuencia de tres nucleótidos denominada PAM3 en la región adyacente a la secuencia diana (Mojica, Díez-Villaseñor, García-Martínez y Almendros, 2009). El corte de la doble hebra de ADN es reparado por la maquinaria celular mediante la eliminación e introducción aleatoria de nucleótidos que generan sitios de apareamiento nuevos entre las dos hebras. Si la reparación se produce en el entorno de la secuencia codificante de un gen, los cambios generalmente conllevan su inactivación (Figura 2). Un segundo mecanismo permite la reparación del corte a partir de un ADN molde en un proceso de recombinación homóloga (Figura 2). Esta segunda vía hace posible la modificación de secuencias específicas que podría tener aplicaciones en las ciencias biomédicas como estrategia de terapia génica para el tratamiento de enfermedades congénitas (Montoliu, 2019).
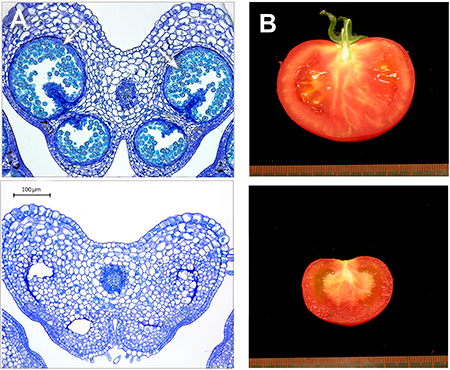
Figura 3. Obtención de plantas de tomate sin polen mediante la tecnología CRISPR/Cas9. La planta editada (abajo) presenta la inserción de un nucleótido en la secuencia de un gen de tomate necesario en el proceso de gametogénesis masculina. A) Cortes histológicos de anteras de flores de tomate. En las plantas convencionales (arriba) se observa la presencia del polen (flechas), mientras que las plantas editadas (abajo) son estériles y no producen polen. B) Frutos maduros de un tomate de la variedad Moneymaker convencional (arriba) y de una planta editada (abajo). En las plantas editadas se obtienen frutos sin semillas (partenocárpicos). / Fotos de Mª Jesús López Martín
En 2012 el trabajo de dos laboratorios propuso la fusión de los dos ARN en una única molécula denominada sgRNA (single-guide RNA) y demostró que es funcional y dirige a la proteína Cas9 a la zona del genoma donde se encuentra el sitio a editar (Gasiunas, Barrangou, Horvath y Siksnys, 2012; Jinek et al., 2012). Los autores también predijeron el potencial de este sistema como herramienta para la edición genética programable mediado por nucleasas (Jinek et al., 2012). No pasaría mucho tiempo hasta la aparición de las primeras publicaciones de edición de genes de organismos in vivo. En 2013 se publican los primeros artículos describiendo la edición de genes en células de mamíferos (Cong et al., 2013) y plantas (Shan et al., 2013).
Desde entonces la utilización de esta tecnología en el editado de genes de diferentes organismos ha tenido un incremento exponencial. Diversos estudios han demostrado que utilizando una sola nucleasa y varios sgRNA se pueden editar varios sitios del genoma simultáneamente, lo cual es especialmente interesante cuando se pretende editar genes de gran tamaño o familias de genes (Wolter, Schindele y Puchta, 2019). La edición de genes en plantas a menudo requiere una etapa inicial de transformación genética para introducir los elementos del sistema (sgRNA y proteína Cas9) y su posterior eliminación en la siguiente generación. Esto supone una limitación para especies en que no se han desarrollado protocolos eficientes de transformación genética o con largos tiempos de generación, como algunas plantas leñosas. En este sentido se están desarrollado métodos alternativos que no requieren transformación estable utilizando protoplastos o embriones inmaduros (Liang et al., 2017; Osakabe et al., 2018). También se han desarrollado sistemas de edición compuestos únicamente por proteínas o complejos RNA-proteína que permiten editar el genoma sin el uso de ADN recombinante (Metje-Sprink, Menz, Modrzejewski y Sprink, 2018).
El futuro de los alimentos y la edición genética en plantas
En plantas de cultivo, el sistema CRISPR/Cas9 se ha utilizado en la mejora de cereales como maíz, arroz y trigo; hortícolas –tomate, patata, pepino y sandía– y especies leñosas –cítricos, papaya y manzano, entre otros– (revisado por Wang, Zhang y Zhu, 2019; Wolter et al., 2019).
La tecnología CRISPR/Cas9 se ha usado extensamente para generar plantas resistentes a estreses tanto abióticos (condiciones ambientales adversas que afectan al crecimiento de la planta) como bióticos (microorganismos patógenos). Recientemente, un grupo del Centro Nacional de Biotecnología en Madrid ha reportado que la edición del gen SlJAZ2 de tomate, relacionado con la percepción de la hormona vegetal jasmonato, confiere protección a la planta frente a hongos patógenos mediante un mecanismo asociado al cierre estomático (Ortigosa, Giménez-Ibañez, Leonhardt y Solano, 2019). En cereales como el arroz, gran parte de las mejoras introducidas mediante la tecnología CRISPR/Cas9 se han concentrado en el desarrollo de plantas más resistentes a hongos fitopatógenos y a incrementar la resistencia a altas temperaturas (Biswal, Mangrauthia, Reddy y Yugandhar, 2019) asociadas a cambio climático.
«La edición genética a la carta tiene un enorme potencial como nueva estrategia en la mejora de los cultivos del futuro»
Un ejemplo de edición genética con importante repercusión en la salud humana es el desarrollo de plantas de trigo con bajo contenido en proteínas del gluten, responsables de diferentes trastornos del sistema digestivo (Sánchez-León et al., 2018). En este trabajo los autores diseñan dos sgRNA para editar regiones conservadas de los genes que codifican las alfa-gliadinas, las principales proteínas que desencadenan sensibilidad en personas que padecen de enfermedad celiaca. La aproximación fue exitosa en la introducción de mutaciones de manera específica y simultánea en esta familia de proteínas, y se obtuvieron plantas con bajo contenido en gluten y una disminución en su inmunoreactividad del 85 % (Sánchez-León et al., 2018). Las líneas obtenidas constituyen un avance sin precedentes en la obtención de variedades comerciales de trigo con bajo contenido en gluten en un futuro cercano.
En nuestro laboratorio del Instituto de Biología Molecular y Celular de Plantas (IBMCP, Valencia) hemos estudiado durante los últimos años el proceso de formación del fruto en plantas de tomate. Nuestros estudios desvelan un mecanismo por el cual las anteras en desarrollo regulan negativamente el crecimiento del ovario no polinizado. En consecuencia, la eliminación temprana de los estambres tiene como consecuencia el desarrollo de frutos sin semillas en esta especie (Medina et al., 2013; Rojas-Gracia et al., 2017).
Basándonos en estos resultados, hemos utilizado la tecnología CRISPR/Cas9 para demostrar que la inactivación específica de genes responsables de la formación del polen es suficiente para desencadenar el desarrollo de frutos sin semillas (partenocárpicos). En concreto, la inserción de un único nucleótido en la secuencia de un gen diana inactiva la función del gen y da lugar a plantas sin polen y partenocárpicas (Figura 3; resultados sin publicar). Este desarrollo podría tener otras aplicaciones en la obtención de plantas ornamentales sin polen como método de contención para evitar su propagación indeseada o para reducir la presencia de alérgenos. Estas nuevas variedades son un ejemplo de edición genética a la carta y demuestran el enorme potencial de estas nuevas estrategias en la mejora de los cultivos del futuro.
Notas
1. Véase la web http://www.theplantlist.org/ (Volver al texto)
2.Las siglas CRISPR/Cas (CRISPER-associated) significan clustered regularly interspaced short palindromic repeats (“repeticiones palindrómicas cortas agrupadas y regularmente espaciadas”). (Volver al texto)
3. Protospacer adjacent motif o “motivo adyacente de protoespaciador”. En el caso de S. pyogenes se ha determinado que la secuencia PAM es NGG, donde N es cualquier nucleótido y G un nucleótido de guanina. (Volver al texto)
Referencias
Beltrán, J. P. (2018). Cultivos transgénicos. Madrid: CSIC-Los libros de la Catarata.
Biswal, A. K., Mangrauthia, S. K., Reddy, M. R., & Yugandhar, P. (2019). CRISPR mediated genome engineering to develop climate smart rice: Challenges and opportunities. Seminars in Cell & Developmental Biology, 96, 100–106. doi: 10.1016/j.semcdb.2019.04.005
Cong, L., Ran, F. A., Cox, D., Lin, S., Barretto, R., Habib, N., . . . Zhang, F. (2013). Multiplex genome engineering using CRISPR/Cas systems. Science, 339(6121), 819–823. doi: 10.1126/science.1231143
FAO. (1983). World food security: A reappraisal of the concepts and approaches. Roma: Food and Agriculture Organization of the United Nations.
FAO. (1999). Women: Users, preservers and managers of agrobiodiversity. Roma: Food and Agriculture Organization of the United Nations.
FAO, FIDA, & PMA. (2012). El estado de la inseguridad alimentaria en el mundo. Roma: Organización de las Naciones Unidas para la Alimentación y la Agricultura.
Gasiunas, G., Barrangou, R., Horvath, P., & Siksnys, V. (2012). Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proceedings of the National Academy of Science of the USA, 109(39), E2579–2586. doi: 10.1073/pnas.1208507109
Jinek, M., Chylinski, K., Fonfara, I., Hauer, M., Doudna, J. A., & Charpentier, E. (2012). A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 337(6096), 816–821. doi: 10.1126/science.1225829
Lander, E. S. (2016). The Heroes of CRISPR. Cell, 164(1-2), 18–28. doi: 10.1016/j.cell.2015.12.041
Liang, Z., Chen, K., Li, T., Zhang, Y., Wang, Y., Zhao, Q., . . . Gao, C. (2017). Efficient DNA-free genome editing of bread wheat using CRISPR/Cas9 ribonucleoprotein complexes. Nature Communications, 8, 14261. doi: 10.1038/ncomms14261
Medina, M., Roque, E., Pineda, B., Cañas, L., Rodríguez-Concepción, M., Beltrán, J. P., & Gómez-Mena, C. (2013). Early anther ablation triggers parthenocarpic fruit development in tomato. Plant Biotechnology Journal, 11(6), 770–779. doi: 10.1111/pbi.12069
Metje-Sprink, J., Menz, J., Modrzejewski, D., & Sprink, T. (2018). DNA-free genome editing: Past, present and future. Frontiers in Plant Science, 9, 1957. doi: 10.3389/fpls.2018.01957
Mojica, F. J., Díez-Villaseñor, C., García-Martínez, J., & Almendros, C. (2009). Short motif sequences determine the targets of the prokaryotic CRISPR defence system. Microbiology, 155(Pt 3), 733–740. doi: 10.1099/mic.0.023960-0
Mojica, F. J., & Montoliu, L. (2016). On the origin of CRISPR-Cas technology: From prokaryotes to mammals. Trends in Microbiology, 24(10), 811–820. doi: 10.1016/j.tim.2016.06.005
Montoliu, L. (2019). Editando genes: recorta, pega y colorea. Las maravillosas herramientas CRISPR. Pamplona: Next Door Publishers.
National Academies of Sciences & Medicine. (2016). Genetically engineered crops: Experiences and prospects. Washington, DC: The National Academies Press.
Ortigosa, A., Giménez-Ibáñez, S., Leonhardt, N., & Solano, R. (2019). Design of a bacterial speck resistant tomato by CRISPR/Cas9-mediated editing of SlJAZ2. Plant Biotechnology Journal, 17(3), 665–673. doi: 10.1111/pbi.13006
Osakabe, Y., Liang, Z., Ren, C., Nishitani, C., Osakabe, K., Wada, M., . . . Nagamangala Kanchiswamy, C. (2018). CRISPR-Cas9-mediated genome editing in apple and grapevine. Nature Protocols, 13(12), 2844–2863. doi: 10.1038/s41596-018-0067-9
Rojas-Gracia, P., Roque, E., Medina, M., Rochina, M., Hamza, R., Angarita-Díaz, M. P., . . . Gómez-Mena, C. (2017). The parthenocarpic hydra mutant reveals a new function for a SPOROCYTELESS-like gene in the control of fruit set in tomato. New Phytologist, 214(3), 1198–1212. doi: 10.1111/nph.14433
Sánchez-León, S., Gil-Humanes, J., Ozuna, C. V., Giménez, M. J., Sousa, C., Voytas, D. F., & Barro, F. (2018). Low-gluten, nontransgenic wheat engineered with CRISPR/Cas9. Plant Biotechnology Journal, 16(4), 902–910. doi: 10.1111/pbi.12837
Shan, Q., Wang, Y., Li, J., Zhang, Y., Chen, K., Liang, Z., . . . Gao, C. (2013). Targeted genome modification of crop plants using a CRISPR-Cas system. Nature Biotechnology, 31(8), 686–688. doi: 10.1038/nbt.2650
Taylor, S. L., & Hefle, S. L. (2001). Ingredient and labeling issues associated with allergenic foods. Allergy: European Journal of Allergy and Clinical Immunology, 56(67), 64–69. doi: 10.1034/j.1398-9995.2001.00920.x
Wang, T., Zhang, H., & Zhu, H. (2019). CRISPR technology is revolutionizing the improvement of tomato and other fruit crops. Horticulture Research, 6(1), 77. doi: 10.1038/s41438-019-0159-x
Wolter, F., Schindele, P., & Puchta, H. (2019). Plant breeding at the speed of light: the power of CRISPR/Cas to generate directed genetic diversity at multiple sites. BMC Plant Biology, 19(1), 176. doi: 10.1186/s12870-019-1775-1