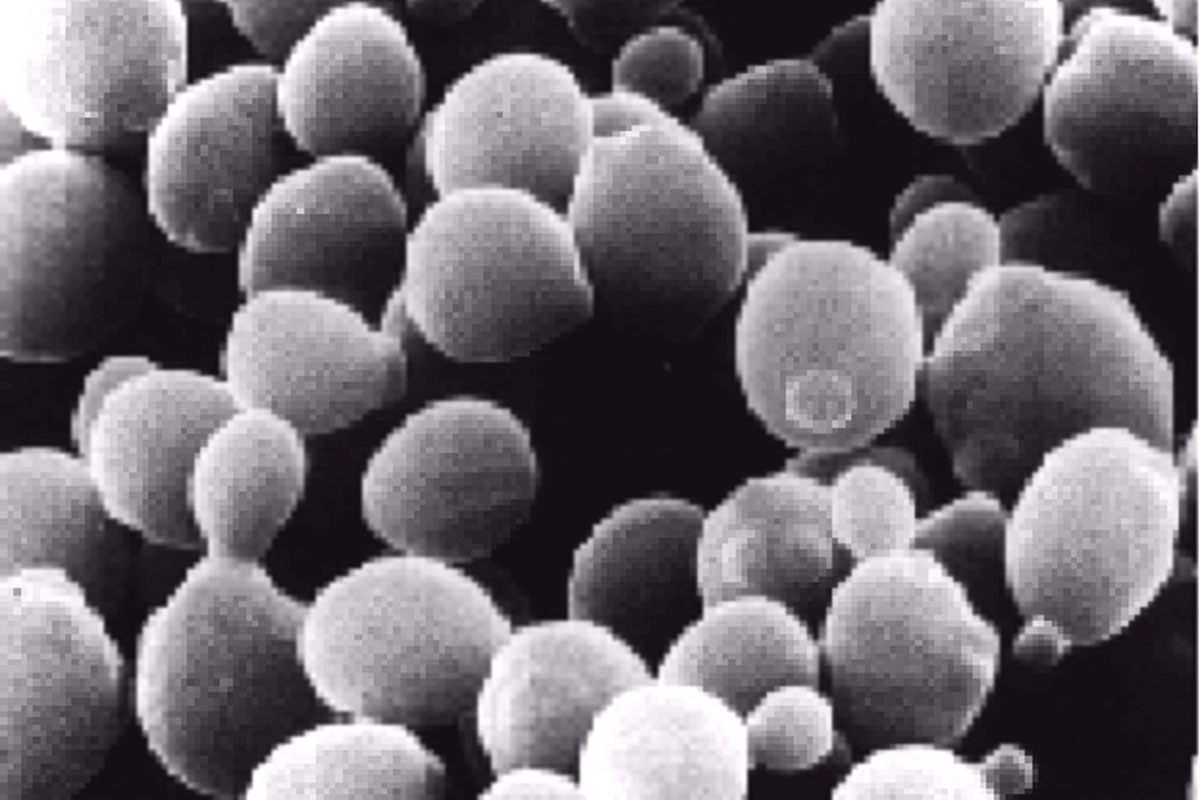
What about the other genomes? 1995 saw the publication of the first complete genome sequences of prokaryotic cells and a year later the first sequences of an eukaryotic organism were published: those of the yeast Saccharomyces cerevisiae. From this moment on, a new scientific discipline came into being related to the study of the genome. This new field of study came into its own, as far as its presence in the press is concerned, with the announcement of the human genome project. Since then, hardly a week goes by without some news item concerning the human genome. The authors question, however, whether this is the only possible genome structure and if the medical applications are the only relevant ones.
Haciendo pan y genómica con las mismas herramientas
En la década de los 80, el desarrollo de las técnicas de secuenciación del DNA llegó a tal nivel de eficacia que en distintos lugares del mundo se empezó a plantear la posibilidad de secuenciar de forma sistemática genomas completos de organismos. En Estados Unidos se lanzó la idea de la secuenciación del genoma humano. En Europa las ideas fueron más modestas, pero quizás más realistas, y se pensó que era preferible secuenciar organismos modelo con genomas más pequeños. La ventaja del enfoque europeo residía en que, con una inversión muy inferior, se podía obtener casi tanta información sobre los genes de un organismo eucariota como la que se pensaba obtener con la secuenciación del genoma humano. Por otro lado, la secuenciación de genomas pequeños podía servir para poner a punto las técnicas necesarias para afrontar proyectos como el del genoma humano, de una envergadura miles de veces superior a cualquier otro proyecto de secuenciación realizado hasta esas fechas. Como antes se indicó, se escogió el genoma de Saccharomyces cerevisiae. Los motivos eran obvios: era el organismo eucariótico mejor conocido, su genoma es pequeño (13 Mb) y compacto, es decir, con muy poco DNA no informativo, se disponía de un elevado número de mutantes, y muchos de sus genes ya estaban secuenciados. El resultado de esta apuesta fue que el genoma de este microorganismo fue el primer genoma eucariota desvelado. Por ello, varios años antes que en cualquier otro organismo, se iniciaron proyectos de genómica funcional que han permitido conocer este ser vivo como ningún otro.
«Quizá lo más atractivo del genoma de la levadura es su gran densidad génica, ya que el 72% del genoma son genes»
Quizás lo más atractivo del genoma de la levadura es su gran densidad génica, ya que el 72% del genoma son genes, lo cual deja muy poco espacio para DNA no codificante y otros elementos funcionales. La longitud media de un gen es de 1.922 pb. La extrapolación de estos datos a todo el genoma de la levadura da un valor de 6.200 ORF predichas. Una cuestión importante es si el número estimado de genes anteriormente descrito es el necesario para el funcionamiento de la célula. De hecho, los estudios de interrupción de genes han demostrado que sólo un pequeño número (15%) de los genes de levadura son esenciales para el crecimiento en medios ricos. Sin embargo, este dato puede ser engañoso, porque muchos pueden desempeñar funciones no esenciales en todo momento, otros pueden resultar sólo esenciales en condiciones de competencia natural entre cepas o con otras especies de microorganismos y, finalmente, otros muchos pueden tener copias más o menos homólogas dentro del propio genoma de la levadura.
Este último dato se ha podido establecer con bastante precisión con el conocimiento de la secuencia completa del genoma. De hecho, un alto porcentaje del genoma es redundante. Se han encontrado casos de repeticiones de genes casi (o totalmente) idénticos, casos de homólogos parciales con funciones iguales o distintas y finalmente, duplicaciones de regiones enteras de cromosomas. La duplicación de buena parte de los genes y, en cierta manera, de su ordenación en los cromosomas ha sugerido que el genoma de Saccharomyces cerevisiae es el resultado de una antigua duplicación de todo el genoma en un tiempo lejano y una posterior evolución con pérdida de parte de los genes duplicados y algunas reorganizaciones cromosomales. Aunque, de acuerdo con lo dicho anteriormente, parece claro que un cierto porcentaje de la redundancia génica es, en realidad, sólo aparente, también podría ser útil para la célula el que ciertos genes estén completamente duplicados en cuanto a las funciones. De esa forma se dispondría de un cierto reservorio de genes repetidos que impedirían que mutaciones puntuales o deleciones producidas por recombinaciones entre cromosomas fueran peligrosas para la célula. Por otro lado, la duplicación de genes es un bien conocido método de evolución para la adquisición de nuevas funciones sin perder las antiguas y su existencia en levadura puede ser una prueba de que los eucariotas utilizan profusamente este medio para adaptarse evolutivamente al medio.
«La genómica puede ayudarnos a saber cuánto de bueno o adecuado para nuestra salud es aquello que comemos»
En cuanto a la función de los genes, sólo un 30% de ellos han sido caracterizados por métodos convencionales y aproximadamente otro 30% tienen homólogos en levadura o en otros organismos con funciones conocidas, por lo que se puede suponer que conocemos también su función (aunque se han producido algunas sorpresas en este tema). Queda un 30-35% de genes que, o bien tienen homólogos de función desconocida (“parejas de huérfanos”) o no tienen homología ni función conocida (“huérfanos solitarios”). En algunos de estos casos, los ordenadores pueden dar cierta idea sobre ellos, pues se puede predecir con cierta seguridad la existencia de hélices transmembrana, dedos de zinc, sitios de fosforilación, etc. Sin embargo este tipo de predicciones no aportan más que algunas pistas, no siempre en la dirección correcta, al problema de encontrar la función de un gen. No obstante, cabe preguntarse si los genes huérfanos tienen alguna característica que los haya hecho “invisibles” al genético o al biólogo molecular. La respuesta no está clara por el momento, pero seguramente la razón de su existencia no es otra que el hecho de que todavía estemos muy lejos de saber cómo funciona una célula y quedan todavía muchas funciones vitales insospechadas por descubrir. En este sentido, sin ningún género de dudas, el genoma del ser vivo con el que hacemos pan, cerveza y vino, la levadura S. cerevisiae, seguirá siendo una potente herramienta que utilizar para aprender cómo funciona la vida.

El coneixement de les interaccions entre dieta i salut arribarà al nivell de les interaccions moleculars. Gràcies a la genòmica funcional podrem conèixer com determinats nutrients afecten l’expressió d’uns gens en concret del nostre organisme.
La secuenciación de genomas procariotas: una herramienta para conocer cuántos genes son necesarios para sustentar la vida
Sin duda, el objetivo final de cualquier proyecto en biología debe ser conocer más y mejor sus bases. En este sentido una pregunta fundamental es: ¿cuál es el tamaño mínimo de un genoma para definir un organismo vivo? La respuesta no es obvia, pero con el advenimiento de un número creciente de genomas secuenciados hemos empezado la comparación sistemática de los genes homólogos ancestrales que tales genomas comparten. De hecho, se ha pensado que éste podría ser un criterio adecuado, aunque hay otros, para aproximarnos al número de genes capaces de sustentar la vida.
El resultado de la comparación de genomas representativos de eubacterias, arqueas y eucariotas, las tres grandes agrupaciones de seres vivos, arroja un número de 80 genes, claramente insuficiente. Son varias las razones que pueden justificar la obtención de un número tan pequeño. Una de ellas es la pérdida de la señal de homología. Al comparar genes de organismos muy alejados en el tiempo evolutivo, cabe la posibilidad de que no podamos determinar si son homólogos. De hecho, el número aumenta cuando comparamos organismos más próximos. Es el caso de las bacterias Mycoplasma genitalium y Ureaplasma urealyticum, dos de los patógenos bacterianos con tamaños genómicos más pequeños (580 kb y 750 kb, respectivamente), que comparten 324 genes. No obstante, cabe señalar que sus adaptaciones específicas, y más concretamente las relacionadas con la capacidad de evadir la respuesta inmune del hospedador, diferente en ambas especies, lleva a identificar la presencia de un número notable de genes derivados, propios de cada una de ellas. Como puede observarse, no resulta sencillo establecer por estos y otros estudios el citado número mínimo.
Ahora bien, unas bacterias particularmente interesantes son aquellas que viven en simbiosis con otros organismos, normalmente de mayor complejidad (protozoos, plantas, animales). Contrariamente a las bacterias patógenas, las bacterias simbiónticas coexisten en forma mutualista con su hospedador, suministrándole productos esenciales, al tiempo que éste les proporciona un ambiente estable así como determinado tipo de metabolitos. De forma exhaustiva se están investigando en la actualidad las bacterias que viven en simbiosis intracelular con los insectos. Una de ellas es Buchnera sp., simbionte de los pulgones. Tiene una propiedad particularmente relevante por lo que hace al número de genes: se trata de un genoma extremadamente reducido, del orden de las 650 kb. Es más, algunas especies del género Buchnera presentan genomas con tamaños inferiores al de M. genitalium. Cabe resaltar el caso de Buchnera sp. de los pulgones que viven sobre pinos y abetos, y cuyo genoma está por debajo de las 500 kb. Concretamente el tamaño del genoma de Buchnera sp. del pulgón Cinara cedri es de tan sólo 450 kb, el menor de los detectados hasta el momento. Pues bien, al igual que se han llevado a cabo estudios orientados a la determinación de los genomas mínimos por comparación de los genomas de bacterias patógenas con genoma de tamaño reducido, estamos en condiciones de hacer lo mismo con algunas bacterias simbiónticas, algunos de cuyos genomas ya se han secuenciado o se están secuenciando, e inferir el número de genes ancestrales compartidos. Otra peculiaridad de las bacterias simbiónticas es que no parecen haber experimentado más que reducción genómica; no han adquirido nuevos genes, como parece haber ocurrido con los genomas de bacterias patógenas, que al menos han ganado aquéllos relacionados con la patogenicidad. Estudios preliminares llevados a cabo con genomas de bacterias simbiónticas, concretamente las especies del género Buchnera anteriormente citadas, y asumiendo genes de un tamaño promedio de 1 kb, nos indican que las mismas probablemente compartan del orden de 330 genes homólogos ancestrales, como cota superior.
El resultado previsible es esperanzador si lo comparamos con aquellos otros obtenidos con las bacterias patógenas de genoma reducido ya mencionadas. De forma independiente, ambos grupos, patógenos y simbiontes con genomas reducidos, parecen tener un número similar de genes compartidos, lo que no quiere decir que sean los mismos genes. El número mínimo podría ser similar, pero la composición es diferente. ¿Qué mensaje se oculta tras está información? Todavía no lo sabemos, pero es evidente que la genómica de estos procariotas nos ayudará a entenderlo.
Cuando la genómica enseña a comer
La industria agroalimentaria está atenta a los desarrollos de la genómica. Sin duda, ésta es un área de investigación donde confluirán muchos abordajes distintos que impliquen el estudio de los genomas de diferentes organismos y materias primas implicadas en la elaboración de alimentos (animales de granja, vegetales comestibles, microorganismos utilizados en la fermentación de alimentos y bebidas), en su contaminación, o en la aplicación de los datos del genoma humano a la alimentación.
«La genòmica va a ayudarnos a entender cómo las dietas afectan a nuestra salud. Por ello hay quien ya empieza a hablar de «alimentación al pasaporte genético». ¿Ciencia-ficción o realidad? El tiempo nos lo dirá»
Hay proyectos de secuenciación de varios genomas de vegetales comestibles. Muchos de ellos los llevan a cabo compañías privadas, lo que hace necesaria una reflexión sobre la necesidad de aporte de capital público en este tipo de investigaciones. No es un hecho trivial, ya que los conocimientos adquiridos a través de estos proyectos, junto con los generados por la genómica funcional y la proteómica, serán la base para mejorar los cultivos por técnicas de ingeniería genética. Un ejemplo interesante a este respecto lo suministra el aroma de la fresa. Sabemos que está producido por más de 300 compuestos, entre ellos más de 100 ésteres. Hay por lo tanto muchos genes implicados en este aroma. Pues bien, recientemente y gracias al uso de estrategias de genómica funcional se han identificado un buen número de ellos, lo que sin duda permitirá en un futuro próximo mejorar esta característica organoléptica.
Se han secuenciado los genomas de una variedad de microorganismos patógenos responsables de toxiinfecciones alimentarias, como por ejemplo Escherichia coli O157:H7, Campylobacter jejuni, Staphylococcus aureus o Listeria monocytogenes. De los datos obtenidos ha sido posible inferir las bases moleculares que permiten a algunos de estos microorganismos sobrevivir en condiciones extremas, abriendo la posibilidad de diseñar procesos industriales de eliminación más eficaces. De la misma forma se han secuenciado los genomas de varias bacterias responsables de la producción de derivados lácteos como Lactococcus lactis, Lactobacillus acidophilus, Lactobacillus bulgaricus o Streptococcus thermophilus, o probióticos como Bifidobacterium breve o Bifidobacterium longum. Muchos de estos proyectos han servido para llevar a cabo comparaciones in silico y determinar qué genes son importantes para llevar a cabo los procesos metabólicos de interés industrial. Y como antes se indicó, en el caso de la levadura Saccharomyces cerevisiae se dispone de la secuencia y, desde hace unos años, de datos de genómica funcional que indican, por ejemplo, qué genes se expresan a lo largo de una fermentación vínica. En resumen, durante los últimos años se han obtenido muchos resultados que permitirán abordar de forma racional estrategias de mejora de la calidad organoléptica, sanitaria o nutricional de los alimentos.
Pero no sólo esto. La genómica puede ayudarnos a saber cuánto de bueno o adecuado para nuestra salud es aquello que comemos. Recientemente se ha estudiado en un modelo experimental animal la interacción entre cepas del género Bacteroides y su huésped utilizando una técnica conocida como perfil de transcripción del genoma (algunos autores lo llaman transcriptoma). Los resultados indican cómo estos microbios de nuestro intestino contribuyen al procesamiento de nutrientes. Pero quizás donde más esperanzas tengamos puestas sea en la correlación entre los datos del genoma humano y la nutrición. Desde hace unos pocos meses la llamada nutrigenómica empieza a tomar fuerza, sobretodo, y significativamente, entre las compañías multinacionales del sector de la agroalimentación. El conocimiento de las interacciones entre dieta y salud va a llegar al nivel de las interacciones moleculares y gracias a la genómica funcional vamos a poder conocer cómo determinados nutrientes afectan a la expresión de determinados genes de nuestro organismo.
No sólo eso, podremos conocer la influencia de variaciones genéticas individuales y su respuesta fisiológica a dietas particulares. En otras palabras, la genómica va a ayudarnos a entender cómo las dietas afectan a nuestra salud. Por ello hay quien ya empieza a hablar de “alimentación al pasaporte genético”. ¿Ciencia-ficción o realidad? El tiempo nos lo dirá, pero quizás no tengamos que esperar mucho para tener una respuesta a esta pregunta gracias a la genómica.