El ADN reescribe nuestra memoria
La recuperación de los desaparecidos a través de su perfil genético

Los continuos avances en el análisis del ADN con fines forenses han ido marcando hitos en la búsqueda de la identidad genética en casos de tipo criminal, de catástrofes y de desapariciones. Los adelantos tecnológicos en el estudio del genoma nos permiten inferir quién y cómo era el dueño de unos restos encontrados, por ejemplo, en una fosa común o una tumba anónima: dónde vivió, cómo era físicamente, o cuál era su origen familiar. El análisis de ADN de interés forense consigue, gracias a una serie de variaciones fijas entre individuos, identificarlos individualmente mediante su perfil genético. Esta identificación puede realizarse por comparación de los perfiles del resto humano con perfiles conocidos o por su compatibilidad con los heredados de sus familiares.
Palabras clave: personas desaparecidas, perfil genético, perfil de metilación, perfil fenotípico, perfil biogeográfico.
Recuperando el pasado
Desde el descubrimiento de la huella genética por el grupo de Alec Jeffreys, de la Universidad de Leicester, en 1985, se han sucedido continuos progresos en el análisis del ADN con fines forenses, tanto a nivel tecnológico como en el tipo de marcadores genéticos analizados y en la estandarización de estos. Gracias al esfuerzo de muchos grupos de investigación y de trabajo de la Sociedad Internacional de Genetica Forense (Bär et al., 1997; Bodner et al., 2016; Carracedo et al., 2000; Mundorff, Bartelink y Mar-Cash, 2009; Parson et al., 2016), estos avances han ido marcando hitos en la búsqueda de la identidad genética para el esclarecimiento de causas criminales, la gestión de grandes catástrofes (Prinz et al., 2007) o la investigación de desapariciones.
«Los avances tecnológicos en el estudio del genoma nos permiten inferir quién y cómo era el dueño de unos determinados restos»
El ADN no puede verse fácilmente, por eso ha pasado desapercibido durante tantos años, pero puede leerse siempre, y cada vez con más claridad, en aquellos restos humanos olvidados por la transmisión familiar y cultural, cuya historia podremos recordar o completar a través de su huella genética. Los avances tecnológicos en el estudio del genoma nos permiten inferir quién y cómo era el dueño de unos determinados restos: dónde vivió, cómo era físicamente, o cuál era su genealogía (Figura 1). Y con el avance de la genómica podremos deducir otros muchos aspectos de la identidad social que, hoy por hoy, solo podemos concebir con la imaginación.
Sabemos que no hay historia sin memoria: por eso siempre hay constancia del éxito y la victoria y, en cambio, debemos esforzarnos para recomponer el fracaso y la vergüenza de nuestra especie. No obstante, si ya somos capaces de leer nuestra prehistoria genómica neandertal (Green et al., 2010; Noonan et al., 2006) y de otros ancestros extinguidos hace miles de años, una prehistoria larvada en las líneas de nuestro propio ADN (Dannemann y Kelso, 2017), pronto seremos capaces de recuperar la memoria de aquellas personas cuyos restos se olvidaron o se ocultaron en el siglo pasado, fuera en periodo de guerra o de postguerra. Del mismo modo, pronto seremos capaces de rescatar a los que están siendo víctimas del olvido en el presente, víctimas de los numerosos conflictos bélicos actuales y de las oleadas de migrantes que estos generan, además del hambre, la discriminación y la injusticia institucionalizada.
La tecnología continúa avanzando, mejorando el manejo de los materiales que hemos de estudiar en la identificación de los restos de seres queridos exhumados de fosas comunes o de tumbas anónimas, o de aquellos recuperados de las playas del Mediterráneo. De igual manera, cada día mejoran los métodos de extracción, restauración y lectura de la secuencia genómica. Así, no solo podemos hacer análisis de parentesco o comparar los resultados con muestras indubitadas de una víctima, sino que incluso somos capaces, en ocasiones, de obtener algunos de sus rasgos fenotípicos o información sobre el origen de sus ancestros, y establecer una aproximación probabilística de su edad o de algunas características de su estilo de vida. Sin embargo, no quisiéramos dar la impresión de que es muy sencillo establecer la identidad de unos restos humanos. Devolverles el nombre y el apellido a partir de la información indirecta aportada por el estudio científico no siempre es posible, ya que muchas veces están tan degradados que no pueden contarnos lo que esperamos conocer: su identidad individualizada.
En los muros de una ciudad se podía leer «sin memoria no hay aprendizaje»: quien hace memoria está apostando por el futuro, por conservar su identidad y su rigor, lo que le permitirá sobrevivir a la famosa pátina del tiempo. Sin memoria no podemos atesorar conocimiento, ni aprender de los aciertos y errores de épocas pasadas, ya sea a través de sus huellas o de sus restos, que nos narran la historia de lo que sucedió y que no conocemos. La buena historia ha de hacerse como una suma de las verdades: las de los héroes y los próceres, pero también las de los testigos anónimos u ocultos. A estos, hay que darles una identidad, para que podamos aprender sobre el momento en que vivieron, soñaron o desaparecieron. En esta labor andan empeñados historiadores, artistas, paleontólogos, antropólogos y, desde hace unas pocas décadas, los genetistas.
Tras estas reflexiones intentaremos explicar en este texto, gracias a los avances en la tecnología, qué preguntas podemos hacer a esos restos, de cuáles tendremos una pronta respuesta y para cuáles habrá que esperar todavía.
Mirando el presente
Desde la secuenciación completa del genoma humano a principios del siglo XXI (Venter et al., 2001) hasta la fecha, se han desarrollado múltiples técnicas que permiten mejorar el análisis de ADN de interés forense, empleado para colaborar en la identificación de los restos de desaparecidos, tanto en conflictos armados como en catástrofes o trata de personas.
Este «tipado» de ADN se consigue gracias al esclarecimiento de la variación genética entre humanos: nuestro genoma presenta una serie de variaciones fijas entre sujetos que permiten identificarlos individualmente. Esta identificación puede realizarse por comparación de los perfiles de los restos humanos con perfiles conocidos o por su compatibilidad con la herencia de progenitores identificados, como ocurre cuando realizamos la investigación forense más básica: la investigación biológica de la paternidad.
En este caso, comparamos el perfil genético del hijo con el de su madre (de la que usualmente no se duda, ya que existe documentación del parto). La variación genética entre ambos genomas, es decir, aquello que no ha heredado el hijo de la madre biológica, ha de provenir de forma obligatoria del progenitor masculino o padre biológico. Este tipo de método también se aplica en los casos de desaparecidos, en los que compararemos la información genética de los familiares más idóneos con la del desaparecido (ya sea vivo o a partir de sus restos), confrontando la herencia de esa información de forma «neutra» –en esta circunstancia, dudamos de ambos padres– ya que no tenemos la certeza de que esos restos sean o no del familiar desaparecido.

Figura 2. Un perfil genético de marcadores tipo STR es como un código de barras propio de cada persona. En la imagen, observamos un perfil de este tipo obtenido mediante análisis de fragmentos de ADN de un individuo anónimo en un secuenciador automático. Los colores corresponden a cada uno de los marcadores fluorescentes empleados y cada pico de la imagen del electroferograma representa una variante (alelo) del individuo analizado. Para elaborar este perfil, se ha empleado un kit comercial para analizar 17 microsatélites en diferentes cromosomas e incluye el gen de la amelogenina (el primer pico de la fila en azul), que permite conocer el sexo masculino (XY) o, como es el caso de este perfil, femenino (XX). / Mercedes Aler
Este tipo de identificación se realiza técnicamente analizando pequeños fragmentos del genoma, los microsatélites o STR (de las siglas en inglés de short tandem repeats), que darán un código o perfil genético concreto. Se trata de repeticiones de grupos de nucleótidos (en general tetra- o pentanucleótidos) que generan fragmentos de ADN con diferentes longitudes entre ellos y muy polimórficos –esto es, muy variables entre las personas– y que al analizarlos de forma automática en un secuenciador nos proporcionan el perfil genético, una especie de código de barras único y exclusivo de cada persona (Figura 2), excepto para gemelos idénticos (univitelinos), pues en este caso ambos poseen el mismo perfil.
Pero cuando no podemos obtener ese perfil completo por una mala calidad de la muestra de partida (restos deteriorados con ADN muy fragmentado o degradado) o necesitamos más respuestas, podemos ampliar el estudio con otro tipo de polimorfismos muchísimo más abundantes en nuestro genoma como son los SNP (siglas en inglés de single nucleotide polymorphism, “polimorfismos de nucleótido único”) y los indel (mutaciones por inserción o deleción de nucleótidos). Analizándolos aumentamos la información necesaria para resolver muchas de las preguntas que no tienen respuesta con los STR.
«El perfil genético es una especie de código de barras único y exclusivo de cada persona excepto para gemelos idénticos»
Hay millones de estos SNP en diferentes segmentos del genoma, y al ser fragmentos de ADN mucho más pequeños que los STR, es más difícil que se degraden y fragmenten. Este análisis, siendo muy útil, precisa de protocolos de trabajo que exigen más tiempo de procesado y, aunque la tasa de mutación es menor que la de un STR, se necesita estudiar más SNP para obtener la misma información que con los STR (Gill, 2001), ya que son menos polimórficos.
Los indel, considerados también como un tipo de SNP, consisten en polimorfismos bialélicos simples de inserción y deleción de uno o pocos pares de nucleótidos. Son sencillos de analizar y pueden estudiarse por separación de tamaños –mediante el análisis de fragmentos–, como en el caso de los STR. Todos estos polimorfismos se analizan con métodos parecidos, que conllevan la amplificación de múltiples fragmentos a la vez (reacción múltiple), que luego se separan en un secuenciador automático, gracias a que los hemos marcado con un producto fluorescente durante la reacción en cadena de la polimerasa (o PCR en las siglas en inglés de polymerase chain reaction).
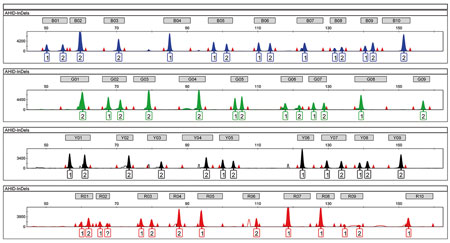
Figura 3. Cuando la mala calidad de una muestra de ADN dificulta la elaboración de un perfil genético completo, el estudio puede ampliarse analizando otros polimorfismos abundantes en nuestro genoma, como los indel, polimorfismos bialélicos simples de inserción y deleción de uno o pocos pares de nucleótidos. En la imagen, podemos observar un perfil genético de 38 marcadores autosómicos de tipo indel para identificación, estudiados mediante análisis de fragmentos de ADN en un secuenciador automático. Cada pico de la imagen del electroferograma representa una variante (alelo) del individuo analizado. / Mercedes Aler
Del análisis de todos ellos se puede obtener un perfil genético específico para cada individuo, con diferente número de lugares del genoma analizados: unos veinte lugares diferentes del genoma para microsatélites o STR, unos cincuenta para un perfil de SNP y unos cuarenta para uno de indel (Figura 3). El número de polimorfismos analizados, del que se obtiene el perfil genético, varía según el producto utilizado para el análisis: los kits comerciales contienen entre diecisiete y veinticuatro y los generados por grupos de investigación incluyen aquellos que cumplen el objetivo concreto de su proyecto (por ejemplo, la elaboración de un perfil fenotípico o ancestral).
En los últimos años, el análisis de variaciones de tipo SNP en el ADN relacionadas con características físicas visibles –el llamado FDP, por las siglas en inglés de forensic DNA phenotyping– está ganando importancia y, en este contexto, podría ser útil cuando queremos conocer información más específica del desaparecido para compararla con la que tenemos de referencia proporcionada por los familiares o allegados (una imagen fotográfica o un documento descriptivo de rasgos, como pueden ser el color de los ojos, de la piel, la forma del cabello…). Es decir, aunque el estudio de marcadores de rasgos fenotípicos no nos sirve por sí mismo para identificar a la persona desaparecida, estos marcadores sí que aportarán respuestas concretas sobre las características físicas que puedan ayudar a identificarla. Así, si los restos que estudiamos coinciden con lo esperado en cuanto a la apariencia y genética de la persona que buscamos, estaremos más cerca de la respuesta.
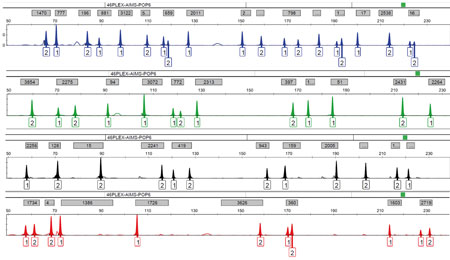
Figura 4. El análisis de los SNP (polimorfismos nucleotídicos simples, en sus siglas en inglés) resulta muy útil, ya que, al tratarse de fragmentos de ADN muy pequeños es más difícil que se degraden y fragmenten. Existen también los marcadores informativos de ancestralidad (AIM en sus siglas en inglés), mediante los cuales puede elaborarse el perfil biogeográfico de un individuo que nos servirá para buscar personas desconocidas que no son identificables con el perfil estándar de ADN. En la imagen, perfil genético de 46 marcadores autosómicos de ancestralidad de tipo indel estudiados mediante análisis de fragmentos de ADN en un secuenciador automático. Cada pico de la imagen del electroferograma representa una variante (alelo) del individuo analizado. / Mercedes Aler
Y, todavía más, con las variantes de los SNP específicos de población denominados AIM (del inglés ancestry informative markers, “marcadores informativos de ancestralidad”) (Figura 4) podemos predecir, mediante la aplicación de potentes parámetros probabilísticos, el llamado «perfil biogeográfico», que nos servirá para buscar personas desconocidas que no son identificables con el perfil estándar de ADN. Este conjunto de marcadores genéticos permite predecir la ascendencia a escala continental e incluso subcontinental, si bien debe tenerse en cuenta que, en la era de la globalización y de las rápidas migraciones humanas en que vivimos, este origen ancestral no tiene por qué coincidir con la nacionalidad (Lipphardt, Toom, Mupepele y Lemke, 2017.
Los marcadores AIM ya se emplean en la actualidad en la resolución de casos criminales en los que, por ejemplo, el perfil genético de una mancha de esperma o de sangre no nos permite la identificación si no tenemos ya el perfil de un sospechoso para compararlos. En estos casos de perfiles «anónimos», la predicción del perfil biogeográfico y del fenotípico puede darnos respuestas acerca de, como ya hemos mencionado, la ascendencia geográfica (europea, asiática, africana…), además del color de los ojos, del cabello y de la piel del donante de la mancha, y proporcionarnos de esta manera información de rasgos del sospechoso anónimo, lo que permite avanzar en la investigación policial. Esto es lo que ocurrió en el caso del crimen de la joven Eva Blanco en 1997, en que el perfil de ADN autosómico y el del cromosoma Y de uno de los investigados sugirió que el hermano de este podría ser el autor del crimen, que pudo resolverse así casi veinte años después mediante el empleo de estos marcadores. A pesar este caso, este tipo de investigaciones mediante el estudio de los AIM no pueden realizarse de forma rutinaria en los laboratorios forenses de España, pues no están al alcance de todos los centros, ni están reguladas de forma específica en nuestra legislación. Cuando se emplean, siempre se hace bajo el mandato de la autoridad judicial.
«El llamado “perfil biogeográfico” nos servirá para buscar personas desconocidas que no son identificables con el perfil estándar de ADN»
Por otra parte, la secuenciación paralela masiva o de nueva generación (NGS en las siglas en inglés de next generation sequencing) está revolucionando, al igual que ocurre con el diagnóstico genético clínico, las aplicaciones forenses del análisis de las variaciones de ADN. Esta técnica, aún en desarrollo para la solución de casos forenses, nos permite analizar muchos y diferentes polimorfismos a la vez (STR, SNP, indel) junto a la secuencia de todas las variantes internas nucleotídicas de los STR, lo cual aumenta su poder de discriminación. Pero la mayor ventaja es la posibilidad de obtener buenos resultados en material altamente degradado porque se pueden analizar fragmentos pequeños con gran profundidad de lectura (es decir, la técnica permite realizar múltiples lecturas sobre la misma muestra, la suma de la cuales permite esa precisión o profundidad). La NGS facilita también el análisis de marcadores de metilación –la huella que deja el paso del tiempo en el ADN– para la predicción de la edad biológica (Abbott, 2018; Freire-Aradas et al., 2016).
Otra aplicación de la NGS es la distinción de gemelos univitelinos a partir de muestras biológicas. Este era uno de los retos de la medicina forense, ya que, en casos criminales, la presunción de inocencia prevalece si no se puede demostrar quién de los dos gemelos pudo cometer el delito. A través de la secuenciación de genomas completos a altas coberturas y detectando mutaciones postcigóticas, tal diferenciación ha dejado de ser un imposible (Vidaki y Kayser, 2017).
Por último, cabe mencionar, por la gran importancia que tiene, el uso de bases de datos poblacionales para obtener los resultados probabilísticos, así como las bases de datos de interés forense, reguladas por ley y en las que se «depositan» perfiles genéticos anónimos, de convictos y de familiares de desaparecidos, todas ellas con interés de servir como identificador si se diera el caso de que fuesen útiles (Ley orgánica 10/2007).
Esperando el futuro
El futuro predecible traerá avances en el campo de la epigenética; sobre todo en la búsqueda de aplicaciones a la existencia constatada de cambios moleculares en las células debidos a la exposición ambiental a lo largo de la vida (Vidaki y Kayser, 2017). El análisis de estas variaciones de origen ambiental en nuestro genoma, ocurridas por la metilación progresiva del ADN, permite obtener –analizando marcadores de tipo CpG– «perfiles de metilación» de interés forense específico en el estudio de restos biológicos escasos y con muy poco ADN, para poder responder a las preguntas siguientes: ¿De qué tipo de tejido es ese resto (sangre, semen, saliva…)? ¿Qué edad biológica tiene el donante? Además, el perfil de metilación podría informar sobre el estilo de vida de un individuo: el consumo de ciertas sustancias (tabaco, alcohol o fármacos), la actividad física, la dieta en las primeras fases de la vida, etc., pues conocemos que todo ello influye en la estructura y apariencia corporal, por lo que, buscando y analizando marcadores específicos, se podrían establecer estos rasgos.
Es evidente, sin embargo, que el uso de estas variaciones implica el esclarecimiento previo de patrones y procesos complejos y variables que suponen un desafío debido a su naturaleza cuantitativa. También se tendrían que superar las barreras tecnológicas que supone el hecho de trabajar con métodos de genotipado múltiple, para poder realizar el análisis cualitativo de varios marcadores simultáneamente. Habrá que identificar los marcadores epigenéticos adecuados, comprobar que se obtienen resultados a partir de una escasa cantidad de ADN inicial, aprender y comprender el alcance de estos, desarrollar modelos estadísticos de predicción y, tan importante como lo anterior, establecer una supervisión y valoración ética y social de los beneficios y riesgos de este tipo de investigaciones.
No querríamos acabar sin mencionar las posibilidades futuras del microbioma (ADN de los microrganismos presentes en un organismo), que podrá aportar desde datos para la identificación personal hasta datos sobre los lugares donde ha vivido el individuo e incluso sus hábitos. Es todavía un campo de estudio en sus comienzos por lo que respecta a sus aplicaciones forenses, pero de grandes posibilidades no exentas de apasionantes dificultades técnicas y teóricas.
Por último, existen planteamientos éticos y legales (Samuel y Prainsack, 2019) pendientes sobre la aplicación de estos avances y de los futuribles, en razón al derecho a la privacidad o al derecho a no saber (Figura 5). Con las técnicas de secuenciación masiva entramos de lleno en el conocimiento de la intimidad genómica y epigenómica con consecuencias posibles en el campo de la salud, del trabajo, del estilo de vida y de la exposición a determinados factores ambientales. Además, debe tenerse en cuenta la importancia de la evaluación e interpretación de los datos y de acotarlos adecuadamente para la aplicación prevista, a fin de evitar errores de interpretación de resultados. Tal como señala la Comisión Internacional sobre Personas Desaparecidas (Parsons, Huel, Bajunović y Rizvić, 2019), estos errores pueden tener consecuencias sociales o judiciales en las identificaciones realizadas en zonas de conflicto bélico o tras grandes catástrofes.
Como ya hemos comentado, todos estos avances precisan de una regulación adecuada, pero, como siempre, la tecnología avanza más rápidamente que el derecho. Así pues, tendremos que comenzar a legislar para ajustar el empleo que hacemos de ella sin afectar a los derechos fundamentales de las personas.
Referencias
Abbott, A. (2018). European scientists seek ‘epigenetic clock’ to determine age of refugees. Nature, 561(7721), 15. doi: 10.1038/d41586-018-06121-w
Bär, W., Brinkmann, B., Budowle, B., Carracedo, A., Gill, P., Lincoln, P., … Olaisen, B. (1997). DNA recommendations. Further report of the DNA Commission of the ISFH regarding the use of short tandem repeat systems. International Society for Forensic Haemogenetics, 110(4), 175–176.
Bodner, M., Bastisch, I., Butler, J. M., Fimmers, R., Gill, P., Gusmão, L., … Parson, W. (2016). Recommendations of the DNA Commission of the International Society for Forensic Genetics (ISFG) on quality control of autosomal Short Tandem Repeat allele frequency databasing (STRidER). Forensic Scientific International: Genetics, 24, 97–102. doi: 10.1016/j.fsigen.2016.06.008
Carracedo, A., Bär, W., Lincoln, P., Mayr, W., Morling, N., Olaisen, B., … Wilson M. (2000). DNA commission of the International Society for Forensic Genetics: Guidelines for mitochondrial DNA typing. Forensic Scientific International, 110(2), 79–85.
Dannemann, M., & Kelso, J. (2017). The contribution of Neanderthals to phenotypic variation in modern humans. American Journal of Human Genetics, 101(4), 578–589. doi: 10.1016/j.ajhg.2017.09.010
Freire-Aradas, A., Phillips, C., Mosquera-Miguel, A., Girón-Santamaría, L., Gómez-Tato, A., Casares de Cal, M., … Lareu, M. V. (2016). Development of a methylation marker set for forensic age estimation using analysis of public methylation data and the Agena Bioscience EpiTYPER system. Forensic Science International: Genetics, 24, 65–74. doi: 10.1016/j.fsigen.2016.06.005
Green, R. E., Krause, J., Briggs, A. W., Maricic, T., Stenzel, U., Kircher, M., … Pääbo, S. (2010). A draft sequence of the Neanderthal genome. Science, 328(5979), 710–722. doi: 10.1126/science.1188021
Gill, P. (2001). An assessment of the utility of single nucleotide polymorphisms (SNPs) for forensic purposes. International Journal of Legal Medicine, 114(4-5), 204–210.
Ley orgánica 10/2007, de 8 de octubre, reguladora de la base de datos policial sobre identificadores obtenidos a partir del ADN. (2007). Consultado en
https://www.boe.es/eli/es/lo/2007/10/08/10/con
Lipphardt, V., Toom, V., Mupepele, A.-C., & Lemke, T. (2017). Open letter on critical approaches to forensic DNA phenotyping and biogeographical ancestry. Consultado el 10 de diciembre, de 2018, en http://www.fb03.uni-frankfurt.de/67048840/Lipphardt-et-al-2017---OPEN-LETTER-ON-CRITICAL-APPROACHES-TO-FORENSIC-DNA-PHENOTYPING-AND-BIOGEOGRAPHICAL-ANCESTRY.pdf
Mundorff, A. Z., Bartelink, E. J., & Mar-Cash, E. (2009). DNA preservation in skeletal elements from the World Trade Center disaster: Recommendations for mass fatality management. Journal of Forensic Sciences, 54(4), 739–745. doi: 10.1111/j.1556-4029.2009.01045.x
Noonan, J. P., Coop, G., Kudaravalli, S., Smith, D., Krause, J., Alessi, J., … Rubin, E. M. (2006). Sequencing and analysis of Neanderthal genomic DNA. Science, 314(5802), 1113–1118. doi: 10.1126/science.1131412
Parson, W., Ballard, D., Budowle, B., Butler, J. M., Gettings, K. B., Gill, P., … Phillips, C. (2016). Massively parallel sequencing of forensic STRs: Considerations of the DNA commission of the International Society for Forensic Genetics (ISFG) on minimal nomenclature requirements. Forensic Science International: Genetics, 22, 54–63. doi: 10.1016/j.fsigen.2016.01.009
Parsons, T. J., Huel, R. M. L., Bajunović, Z., & Rizvić, A. (2019). Large scale DNA identification: The ICMP experience. Forensic Science International: Genetics, 38, 236–244. doi: 10.1016/j.fsigen.2018.11.008
Prinz, M., Carracedo, A., Mayr, W. R., Morling, N., Parsons T. J., Sajantila A., … Schneider P. M. (2007). DNA Commission of the International Society for Forensic Genetics (ISFG): Recommendations regarding the role of forensic genetics for disaster victim identification (DVI). Forensic Science International: Genetics, 1(1), 3–12. doi: 10.1016/j.fsigen.2006.10.003
Samuel, G., & Prainsack, B. (2019). Forensic DNA phenotyping in Europe: Views «on the ground» from those who have a professional stake in the technology. New Genetics and Society, 38(2), 119–141. doi: 10.1080/14636778.2018.1549984
Venter, J. C., Adams, M. D., Myers, E. W., Li, P. W., Mural, R. J., Sutton, G. G., … Zhu, X. (2001). The sequence of the human genome. Science, 291(5507), 1304–1351. doi: 10.1126/science.1058040
Vidaki, A., & Kayser, M. (2017). From forensic epigenetics to forensic epigenomics: Broadening DNA investigative intelligence. Genome Biology, 18(1), 238. doi: 10.1186/s13059-017-1373-1