Tras las huellas de nuestro pasado
Paleoproteómica, ¿el inicio de una nueva era?

Por los restos encontrados, se especula con que Gigantopithecus blacki podía alcanzar los tres metros de altura y pesar alrededor de 300 kg. Pero la carencia de fósiles impedía a la comunidad científica saber más sobre este primate gigante extinto. Ahora, gracias a la paleoproteómica, se ha podido comparar la secuencia de proteínas de esta especie con la de otros grandes simios y establecer que Gigantopithecus era un pariente próximo de los orangutanes. Arriba, representación artística de Gigantopithecus blacki. / Ikumi Kayama (Studio Kayama LLC)
En 1935 el antropólogo alemán Gustav von Koenigswald adquirió unos misteriosos dientes en boticas del sur de China. Los dientes, de unos 2,5 centímetros, se utilizaban en la medicina tradicional china y los llamaban «dientes de dragón». Von Koenigswald quedó perplejo por su tamaño desmesurado. Los dientes mostraban rasgos característicos de los primates, pero eran más grandes que los molares de los gorilas, los primates actuales más grandes del planeta. No es sorprendente, pues, que Von Koenigswald denominara la nueva especie Gigantopithecus. A mediados del siglo xx, nuevos hallazgos de fósiles consolidaron esta especie como un linaje de primates que habitó el sudeste asiático desde hace unos dos millones de años hasta hace 100.000 años, con la incertidumbre de hasta qué punto formaba parte de nuestros orígenes. En la actualidad se han encontrado cuatro mandíbulas y cerca de 1.300 dientes. A pesar de que el tamaño del gigante solo se ha podido extrapolar a partir de huesos del cráneo, se supone que Gigantopithecus podía alcanzar los tres metros de altura y pesar alrededor de 300 kilogramos (Zhang y Harrison, 2017), el primate más grande jamás descubierto.
La comunidad científica no pudo aprender mucho más sobre Gigantopithecus debido a la carencia de fósiles como cráneos completos y esqueletos. Sin datos moleculares, como la secuencia de proteínas o de ADN, las relaciones de fósiles y otros organismos solo se pueden investigar mediante comparaciones morfológicas. De hecho, hasta hace pocas décadas nuestro conocimiento sobre la historia de los homínidos se basaba en la comparación de estructuras óseas entre fósiles. A pesar de que los estudios paleontológicos han sido y son capitales para entender el pasado de nuestra especie, tienen ciertas limitaciones a la hora de resolver posiciones filogenéticas o caracterizar en detalle acontecimientos demográficos.
De la paleogenómica a la paleoproteómica
Actualmente, gracias a grandes adelantos tecnológicos, podemos obtener datos moleculares de organismos que vivieron hace decenas de miles de años mediante la secuenciación de su ADN. Esta «nueva habilidad», la de obtener datos moleculares de organismos fosilizados, ha abierto un gran abanico de posibilidades y ha permitido explorar preguntas antes imposibles de responder. Por ejemplo, gracias al estudio del ADN de neandertales y denisovanos, nuestros primos más próximos, sabemos que su linaje divergió del nuestro hace unos 500.000 años (Prüfer et al., 2014). También sabemos que todas las poblaciones humanas fuera de África tienen cerca de un 3 % de ADN neandertal y que algunas poblaciones de Oceanía muestran, además del ADN de neandertal, hasta un 5 % de material genético proveniente de denisovanos (Reich et al., 2010), muy probablemente como resultado del intercambio de genes entre nuestros ancestros.

Entrada de la cueva de Chuifeng, en China, donde se encontraron los restos de Gigantopithecus de los que ha sido posible obtener proteínas para ser analizadas. Su antigüedad, de unos 1,9 millones de años, no permitía extraer ADN. / Wei Wang
Así pues, si podemos obtener el ADN de organismos fosilizados, ¿cómo es que la comunidad científica no secuencia todo lo que encuentra? La respuesta es bastante intuitiva. Imaginen la Tierra hace unos 1,9 millones de años, momento en el que los restos de un o una Gigantopithecus blacki acabaron en una cueva en el sur de China, la cueva de Chuifeng. Quizás el animal murió allí, o bien algún carroñero transportó parte de sus restos hasta la cueva. En poco tiempo, la carne desapareció y el esqueleto empezó a deteriorarse. Pasaron miles de años, miles de lluvias torrenciales, sequías… incluso múltiples glaciaciones. Todo lo que ha quedado en la actualidad es un diente mineralizado, donde cualquier resto de ADN ha sido desmenuzado en una infinidad de fragmentos moleculares.
La respuesta a la pregunta anterior es, pues, que el ADN no perdura eternamente, se deteriora de forma gradual, se rompe y se transforma en otras moléculas, y acaba convirtiéndose en indetectable o ininterpretable para nosotros. Este deterioro se ve acentuado en climas ecuatoriales y tropicales, puesto que las altas temperaturas aceleran el proceso. De hecho, esta es una de las razones por las que la gran mayoría de estudios de ADN antiguo se han focalizado en muestras localizadas bastante lejos de los trópicos. El «récord mundial» de secuenciación de ADN antiguo se encuentra en 560.000-780.000 años: un caballo preservado en permafrost en las zonas más septentrionales del Canadá, en el territorio de Yukon (Orlando et al., 2013). Fuera de estas situaciones excepcionales para la preservación, el ADN normalmente se puede obtener de organismos que murieron hace unas decenas de miles de años. Por lo tanto, a pesar de su gran potencial, el ADN antiguo se ve limitado a abrir ventanas al pasado más reciente.
«El ADN no perdura eternamente, se deteriora de forma gradual y acaba convirtiéndose en indetectable o ininterpretable»
¿Existe alguna esperanza de obtener datos moleculares de Gigantopithecus de la cueva de Chuifeng? Su antigüedad sugiere que no muchas. Cualquier traza de su ADN se deteriora por el paso del tiempo y acaba completamente estropeado por el clima húmedo y cálido del sur de China. Aun así, las células contienen otros tipos de moléculas además del ADN. La mayoría de libros de biología dedican unas páginas al llamado «dogma central de la biología»: el ADN se transcribe a ARN y a continuación este se traduce a proteínas. A pesar de que este modelo es una gran simplificación del flujo real de información genética dentro de una célula, abre un hilo de luz para nuestro propósito: ¿sería posible secuenciar el ARN o las proteínas de un diente de 1,9 millones de años? El ARN es muy parecido al ADN, una secuencia de nucleótidos donde la timina (T) es reemplazada por uracilo (U), así pues, su estabilidad y resistencia al paso del tiempo es equiparable a la del ADN. Pero… ¿y las proteínas?
«Las proteínas parecen reunir todas las condiciones necesarias para estudiar el pasado más lejano, aquel que es inaccesible por el ADN»
Las proteínas son más estables que el ADN. Ambas moléculas son cadenas de pequeñas unidades (nucleótidos y aminoácidos, respectivamente), pero los enlaces entre las unidades de las proteínas, los enlaces peptídicos, son más resistentes. Además, las proteínas se pliegan en estructuras tridimensionales que pueden protegerlas de su deterioro (en cierto modo, se encuentran plegadas sobre sí mismas, fuera del alcance de influencias externas). Por último, pero no por eso menos importante, las proteínas son más abundantes; excluyendo el agua, aproximadamente el 3 % de la masa de una célula es ADN, muy poco comparado con el 50 % de la masa constituida por proteínas. Por tanto, las proteínas parecen reunir todas las condiciones necesarias para estudiar el pasado más lejano, aquel que es inaccesible por el ADN. A pesar de parecer una idea revolucionaria, en los años cincuenta algunos investigadores ya habían documentado la detección de aminoácidos en fósiles. Aun así, la tecnología para secuenciar proteínas antiguas no existió hasta los años 2000, cuando la comunidad científica adoptó la espectrometría de masas, una técnica típicamente utilizada en disciplinas de la física. Un espectrómetro de masas detecta la presencia de diferentes péptidos (fragmentos cortos de una proteína) en una muestra, y lo consigue analizando su masa (los «pesa»). La información de los péptidos puede ser integrada a posteriori en secuencias de proteínas de longitud más elevada.
Lo que nos puede decir un diente
A finales de 2019, un equipo internacional intentó obtener proteínas del esmalte del Gigantopithecus de Chuifeng mediante la espectrometría de masas. El intento fue un éxito (Welker et al., 2019). Teniendo en cuenta «la edad termal» de la muestra (1,9 millones de años ajustados a la temperatura anual de la cueva), son los datos moleculares de mamífero más antiguos nunca obtenidos. El esmalte del Gigantopithecus es rico en seis proteínas diferentes. Mediante la comparación de las secuencias de proteína obtenida con las del resto de grandes simios actuales (chimpancés, bonobos, orangutanes y humanos), se ha podido establecer que Gigantopithecus era un pariente próximo de los orangutanes. Por otro lado, utilizando el reloj molecular (ritmo al que aparecen cambios en la secuencia de una proteína), se cree que los orangutanes y Gigantopithecus compartieron un ancestro común hace unos diez o doce millones de años. Además, la detección de una proteína nada común en el esmalte de otros simios permite hipotetizar sobre las razones que hay detrás de la distintiva morfología dental de Gigantopithecus. Si Gustav von Koenigswald estuviera vivo, a buen seguro quedaría boquiabierto al ver cómo la tecnología ha transformado la biología en menos de cien años.
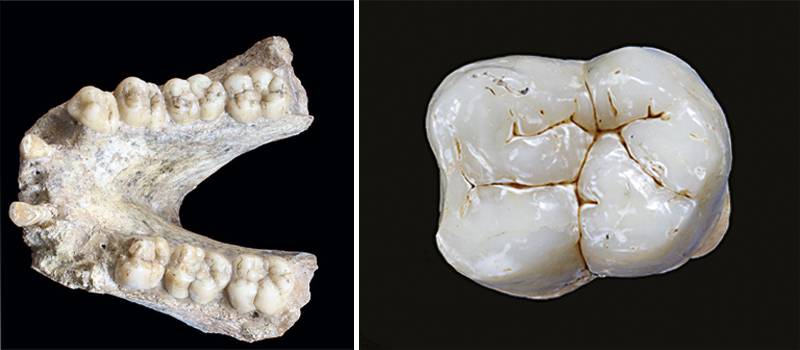
El análisis de la mandíbula de Gigantopithecus blacki ha permitido detectar una proteína nada común en el esmalte de otros simios, lo que permite hipotetizar sobre las razones que existen tras la distintiva morfología dental de esta especie. Este estudio, más allá de los datos que se han podido conocer del gran primate, abre un abanico enorme de nuevas oportunidades para entender nuestro pasado. A la derecha, molar inferior de Gigantopithecus blacki encontrado en la cueva china de Chuifeng. De este esmalte se han podido extraer datos moleculares que hasta el momento son los más antiguos obtenidos jamás de un mamífero. / Wei Wang; Photo retouching: Theis Jensen.
Al margen de ampliar nuestro conocimiento sobre Gigantopithecus, el estudio es importante por razones no directamente relacionadas con el primate gigante. Demuestra que es posible obtener datos moleculares de organismos de millones de años de antigüedad en climas subtropicales. Abre un abanico enorme de nuevas oportunidades para entender nuestro pasado. Un buen ejemplo de estas aplicaciones también lo encontramos en 2019. Los denisovanos son un grupo de homínidos misterioso. Hasta hace poco solo se habían podido identificar a través de cinco fósiles encontrados en la cueva de Denísova (origen de su nombre) en las montañas de Altái, en Rusia. En 2010 se obtuvo la secuencia completa de su genoma (toda la secuencia de ADN de un organismo) a partir de la falange de una niña denisovana (Reich et al., 2010). A pesar de todo eso, nunca se había encontrado un fósil de Denísova fuera de las montañas de Altái. En mayo de 2019, un equipo de investigadores identificó una mandíbula de 160.000 años de antigüedad encontrada en la meseta del Tíbet como denisovana mediante la paleoproteómica (Chen et al., 2019). La antigüedad y el estado de conservación del fósil hacían la extracción de ADN antiguo imposible y, sin la paleoproteómica, probablemente nunca habríamos aprendido que la mandíbula pertenecía a una persona denisovana.
«La paleoproteómica tiene el potencial de llenar los vacíos que todavía hoy existen en el árbol filogenético de los humanos y sus ancestros»
Por otro lado, los casos especialmente interesantes pueden ser aquellos para los cuales no disponemos de datos moleculares. ¿Quiénes eran los misteriosos «hobbits» de la isla de Flores (Homo floresiensis)? ¿Era Homo heidelbergensis nuestro ancestro, o más bien el ancestro de los neandertales? ¿Y Homo erectus? ¿Qué podemos aprender de otro linaje descubierto recientemente como Homo naledi? La lista podría seguir y seguir. La aplicación de la paleoproteómica tiene el potencial de llenar los vacíos que todavía hoy existen en el árbol filogenético de los humanos y sus ancestros cuando nos adentramos más allá de los 500.000 años en el pasado.
A pesar de esta última oda al progreso, el nuevo campo de la paleoproteómica tiene ciertas limitaciones. Aunque las proteínas contienen codificada información genética (tal como describe el «dogma central de la biología»), son menos informativas que el ADN. Principalmente por dos razones. Antes que nada, porque parte de la información genética se pierde en la traducción de ARN a proteína. El código genético es redundante, es decir, que hay diferentes tripletes de ARN que codifican para un mismo aminoácido. Por ejemplo, el aminoácido serina está codificado por dos tripletes de nucleótidos de ARN: AGU y AGC. Así pues, si encontramos una serina en la secuencia de una proteína, no tenemos ninguna herramienta para discernir cuál era el triplete original en el ADN. En segundo lugar, las proteínas son fundamentales para el funcionamiento de una célula, por lo tanto, las regiones de ADN que codifican para proteínas tienen una presión selectiva muy grande, es decir, los cambios de secuencia están muy restringidos, puesto que cualquier modificación podría implicar una pérdida de función. A efectos prácticos, una presión selectiva alta resulta en una baja diversidad de secuencia. Por ejemplo, el colágeno es una proteína que prácticamente tiene la misma secuencia en un humano que en un macaco. Esta observación se explica por la gran importancia del colágeno: millones de años de evolución han generado una secuencia óptima de la proteína, y cualquier mutación será eliminada por el efecto de la selección natural. Por tanto, obtener datos moleculares de proteínas puede resultar en secuencias extremadamente similares entre organismos, lo que dificulta la reconstrucción de filogenias y establecer relaciones evolutivas. Un ejemplo: imaginen que consiguen la secuencia del colágeno de una muestra de Homo floresiensis. Después de dedicar muchísimo tiempo y recursos, se dan cuenta de que su proteína y la nuestra (Homo sapiens) son idénticas, y que, por tanto, no pueden aprender casi nada sobre Homo floresiensis. En resumen, las proteínas pueden llegar a estar tan conservadas en la evolución que obtener la secuencia puede resultar en un ejercicio poco provechoso.
La proteómica también tiene otros retos, en este caso relacionados con las personas y el modus operandi de la comunidad científica. Debe tener cuidado de no caer en la misma trampa que el ADN antiguo hace veinte años. Durante los años noventa y principio de los 2000, ciertas publicaciones defendieron haber obtenido ADN de dinosaurios o insectos atrapados en ámbar (al más puro estilo Jurassic Park), hallazgos que en retrospectiva son difíciles de creer. Más tarde se demostró que estos resultados eran producto de contaminación con ADN moderno o bien de otros errores metodológicos. La paleoproteómica es un campo fascinante que acaparará mucha atención en los próximos años. Así pues, la comunidad científica tendrá que establecer una lista de buenas prácticas, reproducibilidad y transparencia si quiere evitar caer en los mismos errores del pasado.
Sin ningún tipo de duda, nos encontramos en una etapa alentadora para la biología evolutiva. La paleoproteómica tiene el potencial para explorar intervalos de tiempo que eran completamente inaccesibles hasta ahora. Hay que recordar que esta técnica no se ve restringida al estudio de primates o humanos, y puede influir en la investigación en otros animales, plantas, hongos, etc. En 2019 (como pueden ver, un año extremadamente fructífero para la paleoproteómica y posiblemente un augurio de lo que vendrá en el futuro más próximo), se estableció la posición filogenética de un rinoceronte que vivió hace 1,7 millones años (Cappellini et al., 2019). Solo unos pocos años antes, otra investigación obtuvo proteínas de la cáscara de un huevo de avestruz de 3,8 millones de años en Laetoli (Demarchi et al., 2016), el mismo sitio de las famosas huellas de Australopithecus afarensis. Así pues, muy probablemente estamos en el abecé de una oleada de descubrimientos, impulsada por nuevas generaciones de científicos y científicas que quizás cambiarán para siempre jamás nuestra forma de entender la biología evolutiva.
Referencias
Cappellini, E., Welker, F., Pandolfi, L., Ramos-Madrigal, J., Samodova, D., Rüther, P. L., ... Willerslev, E. (2019). Early Pleistocene enamel proteome from Dmanisi resolves Stephanorhinus phylogeny. Nature, 574, 103–107. doi: 10.1038/s41586-019-1555-y
Chen, F., Welker, F., Shen, C.-C. , Bailey, S. E., Bergmann, I., Davis, S., ... Hublin, J.-J. (2019). A late Middle Pleistocene Denisovan mandible from the Tibetan Plateau. Nature, 569, 409–412. doi: 10.1038/s41586-019-1139-x
Demarchi, B., Hall, S., Roncal-Herrero, T., Freeman, C. L., Woolley, J., Crisp, M. K., ... Collins, M. J. (2016). Protein sequences bound to mineral surfaces persist into deep time. eLife, 5, e17092. doi: 10.7554/eLife.17092
Orlando, L., Ginolhac, A., Zhang, G., Froese, D., Albrechtsen, A., Stiller, M., ... Willerslev, E. (2013). Recalibrating Equus evolution using the genome sequence of an early Middle Pleistocene horse. Nature, 499, 74–78. doi: 10.1038/nature12323
Prüfer, K., Racimo, F., Patterson, N., Jay, F., Sankararaman, S., Sawyer, S., ... Pääbo, S. (2014). The complete genome sequence of a Neanderthal from the Altai Mountains. Nature, 505, 43–49. doi: 10.1038/nature12886
Reich, D., Green, R. E., Kircher, M., Krause, J., Patterson, N., Durand, E. Y., ... Pääbo, S. (2010). Genetic history of an archaic hominin group from Denisova Cave in Siberia. Nature, 468, 1053–1060. doi: 10.1038/nature09710
Welker, F., Ramos-Madrigal, J., Kuhlwilm, M., Liao, W., Gutenbrunner, P., De Manuel, M., ... Cappellini, E. (2019). Enamel proteome shows that Gigantopithecus was an early diverging pongine. Nature, 576, 262–265. doi: 10.1038/s41586-019-1728-8
Zhang, Y., & Harrison, T. (2017). Gigantopithecus blacki: A giant ape from the Pleistocene of Asia revisited. American Journal of Physical Anthropology, 162(S63), 153–177. doi: 10.1002/ajpa.23150