¿Se puede estandarizar la vida?
Desafíos actuales en la estandarización de la biología
El concepto de estándar nos remite poderosamente a la idea de máquinas, industrias, dispositivos eléctricos o mecánicos, vehículos o muebles. De hecho, nuestra civilización tecnológica no sería posible –al menos en los términos en los que está estructurada hoy en día– sin componentes universales y fiables cuyo uso generalizado permite alcanzar costes competitivos y asegurar que los productos sean más robustos y sus componentes, más intercambiables. Por ejemplo, un tornillo del Ikea se puede utilizar en toda una serie de muebles estructuralmente diferentes, y una aplicación se puede ejecutar en muchos smartphones distintos. El propio concepto de estandarización está vinculado con la revolución industrial y la producción masiva de bienes en cadenas de montaje. La pregunta que trataremos de responder en el presente documento es hasta qué punto se pueden alcanzar estándares e implementar un proceso de estandarización en el ámbito biológico.
Palabras clave: dependencia del contexto, modularidad, ruido, promiscuidad, biología sintética.

El concepto de estandarización se ha vinculado tradicionalmente con los componentes universales e intercambiables de dispositivos mecánicos o electrónicos, maquinaria, vehículos, muebles, etc. ¿Es posible aplicar la finitud, robustez y universalidad de los estándares al mundo de la biología molecular? / Marco Verch
«El hecho de que tanto los organismos vivos como las máquinas estén sujetos a las leyes de la física no significa que los seres vivos sean máquinas»
La biología sintética, que podemos definir como una aproximación a la biotecnología desde la ingeniería, trata de dotar a la ingeniería genética de un carácter sistemático basado en estándares. La idea central de la biología sintética es hacer más fácil el diseño biológico mediante la incorporación de herramientas y conceptos tomados de la ingeniería (principalmente la industrial y la electrónica). Hoy en día, conceptos de ingeniería como abstracción, desacoplamiento y estandarización se aplican al diseño de sistemas biomoleculares. Estos enfoques aportan nuevos conocimientos sobre las intricadas interacciones moleculares de los sistemas complejos (como las células y las interacciones con su entorno), así como avances en proyectos sobre una gran variedad de cuestiones, desde la agricultura hasta la medicina. No obstante, de este enfoque de ingeniería para la biotecnología surgen muchos desafíos críticos, la mayoría de los cuales quedan fuera del alcance de este texto. Sin embargo, varias secciones de este artículo sí discutirán una de estas cuestiones, la que tiene que ver con algunos retos específicos que plantea el proceso de estandarización en el ámbito biológico, con algo más de profundidad. El hecho de que tanto los organismos vivos como las máquinas estén sujetos a las leyes de la física no significa que los seres vivos sean máquinas (para leer un trabajo exhaustivo y reciente sobre el tema, véase Nicholson, 2019). Las diferencias entre los sistemas complejos producidos por la evolución y los creados por la humanidad están relacionadas con las dificultades para estandarizar los del primer grupo, como discutiremos en las secciones siguientes.
Promiscuidad
Todos los aspectos de la vida son el resultado de una red de interacciones entre diversos componentes, ya sean moléculas intracelulares, las células de los organismos multicelulares o las especies de un ecosistema. Por lo tanto, los seres vivos son sistemas complejos –en el sentido en que se utiliza el término en física–. Por ejemplo, a escala molecular, miles de pequeñas moléculas, macromoléculas y conjuntos supramacromoleculares se ocupan de construir todos los componentes celulares a partir de recursos externos (esto es, se ocupan del metabolismo) o de la capacidad de almacenar y transmitir la información genética (digital) que es necesario descodificar para desarrollar las funciones celulares. Las proteínas son las macromoléculas clave para mantener la actividad biológica, y los bioquímicos y los biólogos estructurales han reunido una impresionante cantidad de detalles sobre la arquitectura, dinámicas e interacciones de estos motores moleculares. En el contexto de la biología sintética, las proteínas se suelen considerar partes intercambiables del sistema que, mediante interacciones moleculares, pueden percibir y procesar las señales y obtener resultados, como ocurre en cualquier sistema de procesamiento de información. Pero los sistemas biológicos de diseño presentan varios problemas que pueden provocar una desviación a partir del comportamiento previsto.
«Los sistemas biológicos de diseño presentan varios problemas que pueden provocar una desviación a partir del comportamiento previsto»
Cuando una proteína concreta se trasplanta a un contexto no nativo, puede provocar un gran abanico de interacciones no deseadas. En los debates sobre los posibles fallos de los sistemas sintéticos y reconstruidos, esta situación se conoce como errores semánticos (Kittleson, Wu y Anderson, 2012). Por ejemplo, la sobreproducción de un metabolito no nativo, como el ácido mevalónico, en Escherichia coli provoca efectos tóxicos como la inhibición de la biosíntesis de ácidos grasos por parte de uno de los intermediarios de la expresión ajena (Kizer, Pitera, Pfleger y Keasling, 2008). Además de la función nativa, esculpida por la selección natural, las proteínas suelen mostrar actividades promiscuas; es decir, reactividades secundarias no deseadas o interacciones que en condiciones normales no representan un daño particular al sistema o son simplemente no adaptativas. Las actividades promiscuas son responsables de lo que se conoce como metabolismo subterráneo (D’Ari y Casadesús, 1998), una colección de reacciones secundarias o menores tradicionalmente pasadas por alto por los bioquímicos, que son el resultado de la habilidad de las enzimas para procesar sustratos distintos a los canónicos (Khersonsky y Tawfik, 2010). Además, se han explotado las promiscuidades moleculares en la evolución in vitro de nuevas actividades enzimáticas, o incluso de nuevas capacidades no naturales, como enzimas que utilizan sustratos o elementos químicos no biológicos (Arnold, 2019). En la naturaleza, las actividades promiscuas también pueden representar un inconveniente si generan productos tóxicos o inhibidores de funciones esenciales. En este sentido, cada vez hay más pruebas de la existencia de muchos sistemas de reparación metabólica –desapercibidos hasta hace poco tiempo– que pueden mitigar o evitar estos problemas de manera eficiente (De Crécy-Lagard, Haas y Hanson, 2018).
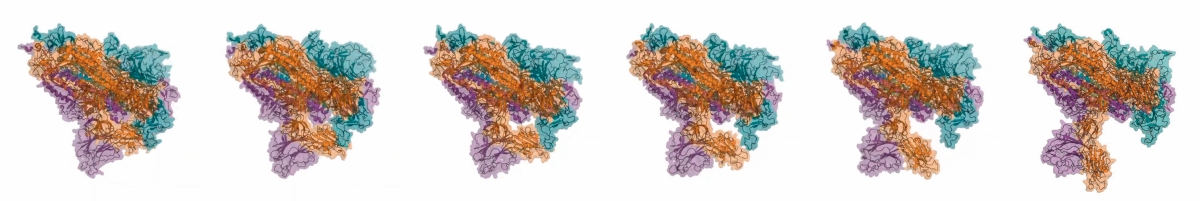
En la biología sintética, las proteínas se consideran partes intercambiables de un sistema que, mediante interacciones moleculares, puede percibir, procesar y transmitir información. Sin embargo, pueden darse interacciones inesperadas en los sistemas biológicos sintéticos, como las actividades promiscuas (esto es, las reacciones e interacciones secundarias, independientes de la función principal de la proteína) que pueden provocar que el sistema funcione de forma incorrecta. Una muestra de cómo la flexibilidad intrínseca de las proteínas pone al descubierto funciones la tenemos en la proteína S del SARS-CoV-2 (en la imagen): el movimiento de uno de sus dominios estructurales expone la superficie de unión con la proteína ACE2 humana. Por lo tanto, uno de los objetivos de la biología sintética debe ser descubrir las funciones desconocidas de muchas enzimas codificadas en genomas y encontrar estrategias para minimizar sus efectos. / FoldingAtHome
En la actualidad, nuestro conocimiento fragmentario sobre las actividades promiscuas supone una grave desventaja para el diseño de novo de los circuitos artificiales. La fusión de proteínas de diferentes orígenes evolutivos en un contexto no nativo puede provocar la acumulación de intermediarios tóxicos o no deseados producidos por las actividades promiscuas. En el futuro, tal vez la biología sintética responda al desafío de los errores semánticos (esto es, de los efectos secundarios inesperados e indeseables) con un esfuerzo conjunto de desarrollo de herramientas para predecir y valorar las actividades promiscuas, actualizando las bases de datos públicas sobre enzimas y proteínas. Tal vez pueda incluso crear un mapa completo de la «materia oscura» metabólica, es decir, descubrir la función desconocida de muchas enzimas codificadas en los genomas (Ellens et al., 2017), así como diseñar estrategias para minimizar las actividades no deseadas mediante el diseño y manipulación de proteínas y células (como, por ejemplo, diseñando complejos supramoleculares artificiales o compartimentos sintéticos que canalicen o almacenen los metabolitos no deseados). El objetivo final de todas estas líneas de trabajo debería ser la descripción detallada de un comportamiento estándar que las biomoléculas de diseño deben seguir al actuar en un contexto no nativo, una situación que contribuirá a la creación de sistemas artificiales más predictivos y optimizados. En cualquier caso, debemos recordar que las proteínas son un tipo de materia blanda y que la promiscuidad es el resultado inevitable de su flexibilidad estructural y comportamiento dinámico intrínsecos. Por lo tanto, es aconsejable aceptar que la inevitabilidad de la promiscuidad de los componentes del sistema es una contingencia en cualquier proyecto de biología sintética, y evaluar su impacto sobre el rendimiento global del sistema de diseño.
«Las funciones biológicas complejas son el resultado de la actividad conjunta de poblaciones de entidades, ya sean moléculas, células o tejidos»
Ruido
Además de la presencia de componentes promiscuos, también tenemos que considerar que las funciones biológicas complejas son el resultado de la actividad conjunta de poblaciones de entidades, ya sean moléculas, células o tejidos. En este sentido, el comportamiento estocástico de los miembros individuales de la población se ha reconocido como una dificultad potencial a la hora de diseñar sistemas biológicos, y como un inconveniente para la estandarización. Incluso en poblaciones celulares genéticamente homogéneas, el comportamiento estocástico de los individuos genera ruido. Esta diversidad funcional se debe a las diferencias y fluctuaciones en la maquinaria molecular disponible dentro de cada una de las células. Incluso en condiciones ideales, dos células pueden contener diferentes cantidades de polimerasas de ARN (enzimas responsables de copiar cada gen en la plantilla correspondiente para ser decodificado), ribosomas (fábricas celulares para la síntesis de proteínas), factores reguladores (que activan o inhiben otros procesos) y otros elementos. Así, cuando las analizamos individualmente, las células muestran variabilidad funcional (Elowitz, Levine, Siggia y Swain, 2002), y este fenómeno también se observa en sistemas diseñados artificialmente. Por ejemplo, uno de los trabajos que se suele considerar fundacional de la biología sintética, el del diseño, simulación e implementación celular de un sistema oscilatorio (el llamado represilador, Elowitz y Leibler, 2000), mostraba variabilidad en las células individuales debido a la estocasticidad de los procesos de expresión génica.
En la naturaleza, el efecto de los fenómenos estocásticos y del ruido puede quedar mitigado por el propio sistema, mientras que en ocasiones se utilizan esos efectos como materia prima para la adaptación. En los sistemas biológicos de diseño se han verificado ambos enfoques (véase Kittleson et al., 2012, y sus referencias). De hecho, en las últimas dos décadas se han implementado con éxito en células algunas operaciones biocomputacionales estándar (puertas lógicas simples) (Amos y Goñi-Moreno, 2018). Sin embargo, queda por responder la pregunta de si sería posible implementar operaciones más complejas dentro de los límites de las células, con su comportamiento y ruido intrínsecos. De forma alternativa, varios grupos han hecho de la necesidad virtud examinando el desorden biológico y los conjuntos multicelulares para comprobar la utilidad de una amplia variedad de enfoques como la computación estocástica, la computación distribuida y la lógica difusa. La última frontera sería trascender el material genético como único soporte para los circuitos lógicos y utilizar otros niveles de operación, como las redes metabólicas, para implementar tareas más complejas (para leer una discusión exhaustiva sobre los logros y retos de la biocomputación, véase Amos y Goñi-Moreno, 2018). En este contexto, sería necesaria una definición más relajada de estándar.

El concurso internacional de biología sintética iGEM es una gran iniciativa para educar a la próxima generación de biólogos sintéticos. A menudo, los ganadores inventan sus propios modelos en lugar de utilizar los estándares validados en trabajos anteriores. En la imagen, el espacio en el que los equipos de estudiantes de todo el mundo muestran sus proyectos a los demás durante el concurso. La imagen corresponde a la edición de 2014. / iGEM/Justin Knight
«Los organismos muestran una increíble gama de variaciones, evidente en los millones de especies que existen»
Dependencia del contexto
Existe un paralelismo entre las variantes de un modelo industrial (un coche, por ejemplo) y las razas o linajes de animales, plantas o bacterias. Los costes económicos de cambiar la maquinaria, las plantillas y los diseños provocan un conjunto relativamente reducido de variaciones de los modelos de coche más populares –o de cualquier otro artefacto–. Por el contrario, los organismos muestran una increíble gama de variaciones, evidente en los millones de especies que existen. Por ejemplo, el organismo modelo microbiológico por excelencia, la bacteria Escherichia coli, incluye miles de cepas con importantes diferencias genéticas (Moradigaravand et al., 2018). Esto nos lleva a la pregunta básica de si todas las cepas se comportan de la misma manera desde el punto de vista de la biotecnología. En una investigación llevada a cabo en un concurso internacional de estudiantes, la competición iGEM, se transformaron seis cepas de E. coli utilizadas habitualmente en biología molecular utilizando un dispositivo simple consistente en una secuencia genética promotora y una reportera; es decir, un circuito genético simple que produce una señal medible, normalmente en forma de luz fluorescente. Mediante la medición de esas señales, se pudo identificar la eficiencia del funcionamiento del circuito en una cepa bacteriana en particular. Sorprendentemente, se encontraron diferencias significativas entre los niveles de expresión de las seis cepas en cinco de los seis dispositivos (Vilanova et al., 2015). En otro experimento del mismo proyecto se transformó una cepa de E. coli utilizando dos circuitos genéticos prácticamente idénticos, uno que codificaba una proteína roja y otro que codificaba una verde. Las células con el fenotipo esperado –a la vez, verde y rojo– resultaron ser rojas, puesto que la mayor parte de la población transformada era roja, verde, o no mostraba fluorescencia (Vilanova et al., 2015). Estos resultados demuestran que la gran variabilidad de cepas o linajes en los organismos vivos implica una falta de reproducibilidad en el comportamiento de componentes genéticos teóricamente «estándar» (entendidos como «universales»). La sensibilidad contextual o la influencia del contexto genético en el rendimiento incluso del circuito de diseño más simple ha sido reconocida como uno de los grandes retos de la biología sintética (Kittleson et al., 2012).
«La gran variabilidad de cepas o linajes en los organismos vivos implica una falta de reproducibilidad en el comportamiento de componentes genéticos teóricamente “estándar”»
El concurso iGEM que hemos mencionado anteriormente representa un esfuerzo espectacular para educar a la próxima generación de biólogos sintéticos. Estudiantes de grado de todo el mundo participan en el concurso y presentan un proyecto de biología sintética que debe basarse, al menos en parte, en un sistema de clonación llamado «Biobricks™». Debido al éxito de las ediciones de iGEM en la última década, Biobricks se ha comenzado a usar de forma masiva, por lo que resultan ideales para realizar un metanálisis sobre la reutilización de componentes en biología sintética. El análisis de los equipos ganadores muestra, no obstante, que la mayoría de ellos tienden a desarrollar sus propios «estándares», en lugar de utilizar los que ya están disponibles –validados, en teoría, por los trabajos anteriores (Vilanova y Porcar, 2014)–. Esto nos lleva a la misma conclusión irónica expresada previamente por el premio Nobel Murray Gell-Mann: «Un científico preferiría utilizar el cepillo de dientes de otra persona antes que la nomenclatura de otro científico» (Vilanova y Porcar, 2019).
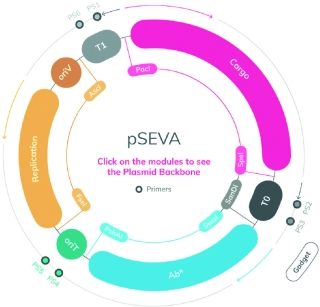
SEVA es un repositorio de plásmidos estandarizados desarrollado en el Centro Nacional de Biotecnología. En la imagen, mapa interactivo que muestra la organización de los vectores SEVA, tal y como aparecen en su versión web 3.0. Cada plásmido contiene tres módulos básicos: una carga o porción de ADN (en magenta), un origen de replicación (en amarillo) y un marcador de antibiótico (en azul). Estos plásmidos pueden usarse para reconstruir y deconstruir fenotipos bacterianos complejos. / SEVA/CNB-CSIC
Dicho esto, hay otros ejemplos de componentes biológicos que se están convirtiendo en estándares de facto en la comunidad de biología sintética. La plataforma Standard European Vector Architecture (SEVA) se compone de una serie de plásmidos diseñados específicamente para ser utilizados en una variedad de especies gramnegativas (y ahora también grampositivas). Son gratuitos y han sido desarrollados por el equipo de Víctor de Lorenzo en el Centro Nacional de Biotecnología (CSIC); se han enviado un total de 2.019 plásmidos a 35 países y las versiones SEVA 2.0 y SEVA 1.0 se han citado en 88 y 277 ocasiones, respectivamente (se acaba de lanzar la versión SEVA 3.0, Martínez-García et al., 2020). Esto pone de relieve la importancia de que un componente biológico sea robusto y útil para que la comunidad de usuarios lo adopte como estándar.
Modularidad
El concepto de módulo está conectado con el de estándar. Los dispositivos mecánicos tienen bloques fácilmente reconocibles diseñados para poderse montar, intercambiar o reparar con facilidad. En biología, los módulos son bastante obvios cuando nos referimos a estructuras anatómicas. Por ejemplo, no es incorrecto definir los pulmones como módulos respiratorios, los músculos como módulos motores o de movimiento, o, en el caso de las células eucariotas, considerar las mitocondrias como módulos internos productores de energía. De hecho, los endosimbiontes son uno de los mejores ejemplos de módulos en los sistemas biológicos (Porcar, Latorre y Moya, 2013). No obstante, cuando trasladamos todas esas estructuras, bien definidas y contenidas en términos físicos, a las vías metabólicas (en otras palabras, si dejamos el hardware biológico y nos ocupamos del software), la situación cambia drásticamente, porque el grado de intercomunicación, variabilidad, ruido y promiscuidad enzimática presente en las vías metabólicas desdibuja en gran medida el concepto de módulo. Esto tiene importantes consecuencias para la biología sintética, puesto que la modularidad implica que un sistema complejo se diseñe, construya y repare de manera mucho más sencilla, porque podemos fraccionarlo en partes más simples. Si los módulos no existen o están conectados con demasiada complejidad entre ellos (esto último es lo que ocurre en los sistemas biológicos), el objetivo de estandarizar módulos/bloques/ vías se convierte en un gran desafío.
Conclusión
En resumen, la estandarización en biología es un reto inmenso debido a la complejidad de los sistemas vivos, su variación y diversidad intrínsecas, la promiscuidad y ruido de las interacciones y catálisis de proteínas, la tendencia de los estándares biológicos a depender del contexto y la ausencia de módulos biológicos genuinos. Solucionar estas cuestiones técnicas es solo el primer paso hacia la estandarización, que es, sobre todo, un proceso social en el que los usuarios finales y los responsables políticos consensuan la elección y el uso de unos estándares. Como hemos visto en este trabajo, todavía estamos lejos de alcanzar estándares en el campo de la biología, pero los éxitos parciales (como los plásmidos SEVA) y la magnitud de los beneficios de estandarizar la biología hacen que el esfuerzo valga la pena.
Referencias
Amos, M., & Goñi-Moreno, A. (2018). Cellular computing and synthetic biology. En S. Stepney, S. Rasmussen & M. Amos (Eds.), Computational Matter (pp. 93–110). Cham: Springer.
Arnold, F. H. (2019) Innovation by evolution: Bringing new chemistry to life (discurso de aceptación del Premio Nobel). Angewandte Chemie International Edition, 58(41), 14420–14426. doi: 10.1002/anie.201907729
D’Ari, R., & Casadesús, J. (1998). Underground metabolism. BioEssays, 20(2), 181–186. doi: 10.1002/(SICI)1521-1878(199802)20:2%3C181::AID-BIES10%3E3.0.CO;2-0
De Crécy-Lagard, V., Haas, D., & Hanson, A. D. (2018). Newly-discovered enzymes that function in metabolite damage-control. Current Opinion in Chemical Biology, 47, 101–108. doi: 10.1016/j.cbpa.2018.09.014
Ellens, K. W., Christian, N., Singh, C., Satagopam, V. P., May, P., & Linster, C. L. (2017). Confronting the catalytic dark matter encoded by sequenced genomes. Nucleic Acids Research, 45(20), 11495–11514. doi: 10.1093/nar/gkx937
Elowitz, M. B., & Leibler, S. (2000). A synthetic oscillatory network of transcriptional regulators. Nature, 403(6767), 335–338. doi: 10.1038/35002125
Elowitz, M. B., Levine, A. J., Siggia, E. D., & Swain, P. S. (2002). Stochastic gene expression in a single cell. Science, 297(5584), 1183–1186. doi: 10.1126/science.1070919
Khersonsky, O., & Tawfik, D. S. (2010). Enzyme promiscuity: A mechanistic and evolutionary perspective. Annual Review of Biochemistry, 79, 471–505. doi: 10.1146/annurev-biochem-030409-143718
Kittleson, J. T., Wu, G. C., & Anderson, J. C. (2012). Successes and failures in modular genetic engineering. Current Opinion in Chemical Biology, 16(3-4), 329–336. doi: 10.1016/j.cbpa.2012.06.009
Kizer, L., Pitera, D. J., Pfleger, B. F., & Keasling, J. D. (2008). Application of functional genomics to pathway optimization for increased isoprenoid production. Applied and Environmental Microbiology, 74(10), 3229–3241. doi: 10.1128/AEM.02750-07
Martínez-García, E., Goñi-Moreno, A., Bartley, B., McLaughlin, J., Sánchez-Sampedro, L., Pascual Del Pozo, H., … De Lorenzo, V. (2020). SEVA 3.0: An update of the Standard European Vector Architecture for enabling portability of genetic constructs among diverse bacterial hosts. Nucleic Acids Research, 48(D1), D1164–D1170. doi: 10.1093/nar/gkz1024
Moradigaravand, D., Palm, M., Farewell, A., Mustonen, V., Warringer, J., & Parts, L. (2018). Prediction of antibiotic resistance in Escherichia coli from large-scale pan-genome data. PLOS Computational Biology, 14(12), e1006258. doi: 10.1371/journal.pcbi.1006258
Nicholson, D. J. (2019). Is the cell really a machine? Journal of Theoretical Biology, 477, 108–126. doi: 10.1016/j.jtbi.2019.06.002
Porcar, M., Latorre, A., & Moya, A. (2013). What symbionts teach us about modularity. Frontiers in Bioengineering and Biotechnology, 1, 14. doi: 10.3389/fbioe.2013.00014
Vilanova, C., & Porcar, M. (2014). iGEM 2.0–refoundations for engineering biology. Nature Biotechnology, 32, 420–424. doi: 10.1038/nbt.2899
Vilanova, C., & Porcar, M. (2019). Synthetic microbiology as a source of new enterprises and job creation: A Mediterranean perspective. Microbial Biotechnology, 12, 8–10. doi: 10.1111/1751-7915.13326
Vilanova, C., Tanner, K., Dorado-Morales, P., Villaescusa, P., Chugani, D., Frías, A., ... Porcar, M. (2015). Standards not that standard. Journal of Biological Engineering, 9, 17. doi: 10.1186/s13036-015-0017-9
El trabajo de los autores ha sido financiado por el Ministerio de Ciencia e Innovación y por fondos del FEDER (RTI2018-095584-B-C41) y de la iniciativa Horizonte 2020 de la Unión Europea (BioRobooST: Fostering synthetic biology standardization through international collaboration, Project ID 210491758).